Myc
From Wikipedia, the free encyclopedia
Myc é unha familia de xenes reguladores e protooncoxenes que codifican factores de transcrición. A familia Myc consta de tres xenes humanos relacionados: c-myc (MYC), l-myc (MYCL) e n-myc (MYCN). c-myc foi o primeiro xene que se descubriu nesta familia, debido á súa homoloxía co xene viral v-myc.
Protoncoxene MYC | |
| Identificadores | |
| Símbolo | MYC |
| Símbolos alt. | c-Myc, v-myc |
| Entrez | 4609 |
| HUGO | 7553 |
| OMIM | |
| RefSeq | NM_001354870.1 |
| UniProt | P01106 |
| Outros datos | |
| Locus | Cr. 8 q24.21 |
Protooncoxene MYCL | |
| Identificadores | |
| Símbolo | MYCL |
| Símbolos alt. | LMYC, MYCL1, bHLHe38, L-Myc, v-myc |
| Entrez | 4610 |
| HUGO | 7555 |
| OMIM | |
| RefSeq | NM_005376 |
| UniProt | P12524 |
| Outros datos | |
| Locus | Cr. 1 p34.2 |
Protooncoxene MYCN | |
| Identificadores | |
| Símbolo | MYCN |
| Entrez | 4613 |
| HUGO | 7559 |
| OMIM | |
| RefSeq | NM_005378 |
| UniProt | V |
| Outros datos | |
| Locus | Cr. 2 p24.3 |
No cancro, c-myc a miúdo exprésase constitutivamente (persistentemente). Isto causa o incremento da expresión de moitos xenes, algúns dos cales están implicados na proliferación celular, contribuíndo á formación do cancro.[1] Unha translocación común en humanos que afecta a c-myc é fundamental para o desenvolveento de moitos casos de linfoma de Burkitt.[2] A regulación á alza constitutiva dos xenes Myc tamén se observou nos carcinomas de cérvix uterino, colon, mama, pulmóns e estómago.[1]
Myc considérase unha prometedora diana para fármacos anticancro.[3] Desafortunadamente, Myc posúe varias características que fixeron difícil aplicarlle fármacos, como que calquera fármaco anticancro dirixido a inhibir Myc normalmente necesitará perturbar a proteína indirectamente, como pode ser afectando ao ARNm da proteína en vez de afectar directamente á propia proteína por medio dunha pequena molécula.[4][5]
Ademais, c-myc tamén ten un importante papel na bioloxía das células nais e foi un dos factores Yamanaka oixinais usados para reprogramar células somáticas en células nais pluripotentes inducidas.[6]
No xenoma humano c-myc está localizado no cromosoma 8 e crese que regula a expresión do 15% de todos os xenes[7] por medio da unión a secuencias de caixa de amplificador (E-box).
Ademais do seu papel como factor de transcrición clásico, N-myc pode recrutar histona acetiltransferases (HATs). Isto permítelle regular a estrutura global da cromatina por acetilación de histonas.[8]
Descubrimento
A familia Myc estableceuse despois do descubrimento da homoloxía entre un oncoxene presente no virus aviar da mielocitomatose (v-myc; P10395, do inglés avian virus, myelocytomtosis) e un xene humano sobreexpresado en varios cancros, o Myc celular (c-Myc, do inglés cellular Myc).[Cómpre referencia] Posteriormente, o descubrimento de máis xenes homólogos en humanos fixo que se engadisen á familia n-Myc e l-Myc.[9]
O exemplo que se pon máis frecuentemente de c-Myc como protooncoxene é a súa implicación no linfoma de Burkitt. No linfoma de Burkitt, as células cancerosas mostran translocacións cromosómicas, normalmente entre os cromosomas 8 e 14 [t(8;14)]. Isto causa que c-Myc quede situado augas abaixo da rexión promotora da inmunoglobulina (iG) altamente activa, o que orixina a sobreexpresión de Myc.
Estrutura
Os produtos proteicos dos xenes da familia Myc pertencen todos á familia de factores de transcrición Myc, os cales conteñen os motivos estruturais bHLH (hélice básica-bucle-hélice) e LZ (cremalleira de leucina). O motivo bHLH permite ás proteínas Myc unirse ao ADN, mentres que o motivo de cremalleira de leucina de unión a factores de transcrición permite a dimerización con Max, outro factor de transcrición bHLH.
O ARNm Myc contén un IRES (sitio de entrada ao ribosoma interno) que permite que o ARN sexa traducido a proteína cando a tradución dependente da caparuza 5' (5'-cap) está inhibida, como durante a infección viral.
Función
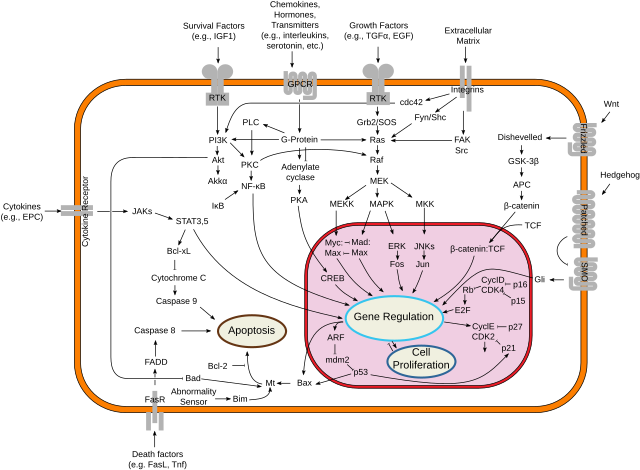
As proteínas Myc son factores de transcrición que activan a expresión de moitos xenes proproliferativos pola súa unión a secuencias box amplificadoras (E-box) e recrutan histona acetiltransferases (HATs). Unha Myc pénsase que funciona regulando á alza a elongación do transcrito en xenes que son activamente transcritos polo recrutamento de factores de elongación transcricional.[10] Pode tamén actuar como represor transcricional. Ao unirse ao factor de transcrición Miz-1 e desprazar o coactivador p300, inhibe a expresión de xenes diana de Miz-1. Ademais, myc ten un papel directo no control da replicación do ADN.[11] Esta actividade podería contribuír á amplificación do ADN en células cancerosas.[12]
Myc é activada por varios sinais mitóxenos, como a estimulación do soro ou por Wnt, Shh e EGF (por medio da vía MAPK/ERK).[13] Ao modificar a expresión dos seus xenes diana, a activación de Myc causa numerosos efectos biolóxicos. O primeiro en ser descuberto foi a súa capacidade de impulsar a proliferación celular (regula á alza as ciclinas, e á baixa a p21), pero tamén xoga un papel moi importante na regulación do crecemento celular (regula á alza o ARN ribosómico e proteínas), a apoptose (regula á baixa Bcl-2), a diferenciación, e a autorrenovación de células nais. Os xenes do metabolismo de nucleótidos están regulados á alza por Myc,[14] e son necesarios para a proliferación inducida por Myc[15] ou crecemento celular.[16]
Fixéronse varios estudos que indicaron claramente o papel de Myc na competición celular.[17]
Un efecto principal de c-myc é a proliferación de células B, e a ganancia de MYC foi asociada con malignidades nas células B e o incremento da súa agresividade, incluíndo a transformación histolóxica.[18] Nas células B, Myc actúa como un oncoxene clásico ao regular varias vías proproliferativas e antiapoptóticas; isto tamén inclúe o afinamento das sinalizacións BCR e CD40 na regulación de microARNs (miR-29, miR-150, miR-17-92).[19]
c-Myc induce a expresión do xene MTDH (AEG-1) e á súa vez el mesmo require o oncoxene AEG-1 para a súa expresión.
Myc-nick

Myc-nick é unha forma citoplasmática de Myc producida por un corte proteolítico parcial das moléculas de lonxitude completa c-Myc e N-Myc.[20] No corte de Myc os mediadores son as proteases citosólicas dependentes de calcio da familia da calpaína.
O corte de Myc feito polas calpaínas é un proceso constitutivo, pero é potenciado en condicións que requiren unha regulación á baixa rápida dos niveis de Myc, como durante a diferenciación terminal. Co corte o C-terminal de Myc (que contén o dominio de unión ao ADN) é degradado, mentres que Myc-nick, o segmento N-terminal de 298 residuos, permanece no citoplasma. Myc-nick contén dominios de unión para as histona acetiltransferases e para as ubiquitina ligases.
As funcións de Myc-nick están investigándose actualmente, pero este novo membro da familia Myc regula a morfoloxía da célula, polo menos en parte, ao interaccionar con acetil transferases para promover a acetilación de α-tubulina. A expresión ectópica de Myc-nick acelera a diferenciación de mioblastos comprometidos en células musculares.
Importancia clínica
Unha gran cantidade de evidencias mostra que os xenes Myc e proteínas son moi relevantes para tratar tumores.[9] Excepto para os xenes de resposta temperán, Myc regula á alza universalmente a expresión de xenes. Ademais, a regulación á alza non é linear. Os xenes para os cales a expresión está xa significativamente regulada á alza en ausencia de Myc son fortemente estimulados en presenza de Myc, mentres que os xenes nos que a expresión é baixa en ausencia de Myc só obteñen un pequeno impulso cando está presente Myc.[6]
A inactivación do encima activador de SUMO (SAE1 / SAE2) en presenza de hiperactivación de Myc ten como resultado unha catástrofe mitótica e morte celular en células cancerosas. Por tanto, os inhibidores da SUMOilación son un posible tratamento para o cancro.[21]
A amplificación do xene MYC atopouse nun número significativo de casos de cancro de ovario epitelial.[22] En conxuntos de datos TCGA, a amplificación de Myc ocorre en varios tipos de cancro, incluíndo o de mama, colorrectal, pancreático, gástrico e uterino.[23]
No proceso de transformación experimental de células normais en células cancerosass, o xene MYC pode cooperar co xene RAS.[24][25]
A expresión de Myc é altamente dependente da función de BRD4 nalgúns cancros.[26][27] Utilizáronse inhibidores BET para bloquear con éxito a función de Myc en modelos de cancro preclínicos e están actualmente sendo avaliados en ensaios clínicos.[28]
A expresión de MYC está controlada por unha ampla variedade de ARNs non codificantes, como o miRNA, ARNncl e ARN circular. Algúns destes ARNs son específicos de certos tipos de tecidos humanos e tumores.[29] Os cambios na expresión de ditos ARNs pode potencialmente usarse para desenvolver unha terapia dirixida a tumores.
Modelos animais
En Drosophila Myc está codificada polo locus diminutivo, (que xa coñecían os xenetistas antes de 1935).[30] Os alelos diminutivos clásicos orixinan un animal viable cun tamaño corporal pequeno. Drosophila foi seguidamente usada en experimentos que implicaron Myc na competición celular,[31] endorreplicación,[32] e crecemento celular.[33]
Durante o descubrimento do xene Myc, comprendeuse que os cromosomas que se translocan reciprocamente ao cromosoma 8 contiñan xenes de inmunoglobulina no punto de rotura. Para estudar o mecanismo da tumoroxénese no linfoma de Burkitt imitando o padrón de expresión de Myc nestas células cancerosas, desenvolvéronse modelos de ratos transxénicos. O xene Myc situado baixo o control do amplificador da cadea pesada de IgM en ratos transxénicos dá lugar a linfomas principalmente. Posteriormente, para estudar os efectos de Myc noutros tipos de cancro, producíronse tamén ratos transxénicos que sobreexpresaban Myc en diferentes tecidos (fígado, mama). En todos estes modelos de ratos a sobreexpresión de Myc causa tumoroxénese, o que ilustra a potencia do oncoxene Myc. Nun estudo en ratos, a expresióin reducida de Myc inducía lonxevidade, con vidas medias e máximas significativamente aumentadas en ambos os sexos e unha taxa de mortalidade reducida en todas as idades, mellor saúde, progresión do cancro máis lenta, mellor metabolismo e corpos máis pequenos. Ademais, presentaban menos TOR, AKT, S6K e outros cambios en enerxía e vías metabólicas (como a AMPK, máis consumo de oxíxeno, máis movementos corporais, etc.). No estudo de John M. Sedivy e outros usouse a Cre-Loxp-recombinase para realizar o knockout dunha copia de Myc e isto resultou nun xenotipo "haploinsuficiente" Myc+/-. Os fenotipos que se observaron opóñense aos efectos do envellecemento normal e son compartidos por moitos outros modelos de ratos de longa vida como o CR (restrición de calorías, calorie restriction) ames dwarf, rapamicina, metformina e resveratrol. Un estudo atopou que os xenes Myc e p53 eran claves para a supervivencia de células de leucemia mieloide crónica. Tomando as proteínas Myc e p53 como dianas de fármacos obtivéronse resultados positivos en ratos con leucemia mieloide crónica.[34][35]
Relación coas células nais
Os xenes Myc desempeñan varias funcións normais nas células nais, incluíndo as céllas nais pluripotentes. Nas células nais neurais, N-Myc promociona as células nais que proliferan raidamente e o estado similar ao precursor no cerebro en desenvolvemento, mentres que inhibe a diferenciación.[36] Nas células nais hematopoéticas Myc controla o balance entre a autorrenovación e a diferenciación.[37]
c-Myc exerce unha importante función na xeración de células nais pluripotentes inducidas (iPSCs). É un dos factores orixinais descubertos por Yamanaka et al. que fomentan que as células volvan ao estado "similar a célula nai" xunto cos factores de tanscrición Oct4, Sox2 e Klf4. Porén, desde entón viuse que é posible xerar iPSCs sen c-Myc.[38]
Interaccións
Myc presenta interaccións con:
- ACTL6A[39]
- BRCA1[40][41][42][43]
- Bcl-2[44]
- Ciclina T1[45]
- CHD8[46]
- DNMT3A[47]
- EP400[48]
- GTF2I[49]
- HTATIP[50]
- let-7[51][52][53]
- MAPK1[44][54][55]
- MAPK8[56]
- MAX[57][58][59][60][61][62][63][64][65][66][67][68][69]
- MLH1[61]
- MYCBP2[70]
- MYCBP[71]
- NMI[40]
- NFYB[72]
- NFYC[73]
- P73[74]
- PCAF[75]
- PFDN5[76][77]
- RuvB-like 1[39][48]
- SAP130[75]
- SMAD2[78]
- SMAD3[78]
- SMARCA4[39][57]
- SMARCB1[60]
- SUPT3H[75]
- TIAM1[79]
- TADA2L[75]
- TAF9[75]
- TFAP2A[80]
- TRRAP[39][58][59][75]
- WDR5[81]
- YY1[82] e
- ZBTB17.[83][84]
- C2orf16[85]
Notas
Véxase tamén
Wikiwand - on
Seamless Wikipedia browsing. On steroids.