Loading AI tools
Template:Chloroplast DNA
Template:Chloroplast DNA
遺伝子マッピングまたはゲノムマッピングは、染色体上の遺伝子の位置と遺伝子間の距離を特定する手法を説明するものである。[2][3] 遺伝子マッピングは、遺伝子内の異なる部位間の距離を説明するものでもある。
Gene mapping or genome mapping describes the methods used to identify the location of a gene on a chromosome and the distances between genes.[2][3] Gene mapping can also describe the distances between different sites within a gene.
ゲノムマッピングの本質は、分子マーカーの集合をゲノム上のそれぞれの位置に配置することである。分子マーカーにはさまざまな種類がある。ゲノムマップの作成においては、遺伝子は遺伝子マーカーの一種として見なされ、他のマーカーと同じ方法でマッピングされる。研究分野によっては、遺伝子マッピングは生物体内における新しい遺伝子組み換え体の作成に貢献している。[4]
The essence of all genome mapping is to place a collection of molecular markers onto their respective positions on the genome. Molecular markers come in all forms. Genes can be viewed as one special type of genetic markers in the construction of genome maps, and mapped the same way as any other markers. In some areas of study, gene mapping contributes to the creation of new recombinants within an organism.[4]
遺伝子地図は、染色体上の遺伝子の空間的配置を記述するのに役立つ。遺伝子は、遺伝子座として知られる染色体上の特定の位置に指定され、染色体上の他の遺伝子間の距離を特定する分子マーカーとして使用することができる。地図は、特定の形質の遺伝パターンを予測する機会を研究者に提供し、最終的には疾患に関連する形質の理解を深めることにつながる可能性がある。[5]
Gene maps help describe the spatial arrangement of genes on a chromosome. Genes are designated to a specific location on a chromosome known as the locus and can be used as molecular markers to find the distance between other genes on a chromosome. Maps provide researchers with the opportunity to predict the inheritance patterns of specific traits, which can eventually lead to a better understanding of disease-linked traits.[5]
遺伝子地図の遺伝的基礎は、DNAの配列決定を研究者が行う際に役立つ可能性のある概要を提供することである。遺伝子地図は、遺伝子の相対的位置を指摘し、研究者がゲノム内の関心領域を特定するのに役立つ。これにより、遺伝子を迅速に特定し、配列決定することができる。[6]
The genetic basis to gene maps is to provide an outline that can potentially help researchers carry out DNA sequencing. A gene map helps point out the relative positions of genes and allows researchers to locate regions of interest in the genome. Genes can then be identified quickly and sequenced quickly.[6]
遺伝子地図(遺伝子マッピング)を作成する2つのアプローチには、物理的マッピングと遺伝的マッピングがある。物理的マッピングでは、分子生物学の技術を用いて染色体を検査する。これらの技術により、研究者は結果的に染色体を直接観察することができ、相対的な遺伝子位置のマップを作成することができる。一方、遺伝子マッピングでは、遺伝学の技術を用いて間接的に遺伝子の関連性を調べる。その技術には、交配(ハイブリッド)実験や家系図の調査が含まれる。これらの技術により、遺伝子やその他の重要な配列の相対的位置を分析するためのマップを作成することができる。[6]
Two approaches to generating gene maps (gene mapping) include physical mapping and genetic mapping. Physical mapping utilizes molecular biology techniques to inspect chromosomes. These techniques consequently allow researchers to observe chromosomes directly so that a map may be constructed with relative gene positions. Genetic mapping on the other hand uses genetic techniques to indirectly find association between genes. Techniques can include cross-breeding (hybrid) experiments and examining pedigrees. These technique allow for maps to be constructed so that relative positions of genes and other important sequences can be analyzed.[6]
ゲノムマッピングの分野では、2つの異なるマッピングアプローチが用いられている。遺伝子地図(連鎖地図とも呼ばれる)[7]と物理地図[3]である。両者とも遺伝子マーカーと遺伝子座の集合であるが[8]、遺伝子地図の距離は遺伝子連鎖情報に基づいており、物理地図は通常、塩基対数で測定される実際の物理的距離を使用している。物理地図はゲノムをより正確に表現できる可能性があるが、遺伝地図はしばしば染色体の異なる領域の性質に関する洞察をもたらす。例えば、遺伝距離と物理距離の比率はゲノムの異なる領域で大きく異なり、これは異なる組み換え率を反映している。そして、こうした率はゲノムのユークロマティック(通常は遺伝子が多い)領域とヘテロクロマティック(通常は遺伝子が少ない)領域を指し示すことが多い。
There are two distinctive mapping approaches used in the field of genome mapping: genetic maps (also known as linkage maps)[7] and physical maps.[3] While both maps are a collection of genetic markers and gene loci,[8] genetic maps' distances are based on the genetic linkage information, while physical maps use actual physical distances usually measured in number of base pairs. While the physical map could be a more accurate representation of the genome, genetic maps often offer insights into the nature of different regions of the chromosome, for example the genetic distance to physical distance ratio varies greatly at different genomic regions which reflects different recombination rates, and such rate is often indicative of euchromatic (usually gene-rich) vs heterochromatic (usually gene-poor) regions of the genome.
Genetic mapping
研究者は、著名な疾患や形質を持つ家族と持たない家族から血液、唾液、組織のサンプルを採取することから遺伝子地図の作成を開始する。遺伝子マッピング、特に個人向けゲノム検査で最もよく使用されるサンプルは唾液である。科学者は次に、サンプルからDNAを分離し、それを詳細に検査して、疾患を持つ家族のDNAに特有のパターンがないか調べる。疾患を持たない家族のDNAにもないパターンである。これらのDNAの分子特有パターンは、多型またはマーカーと呼ばれる。[9]
Researchers begin a genetic map by collecting samples of blood, saliva, or tissue from family members that carry a prominent disease or trait and family members that don't. The most common sample used in gene mapping, especially in personal genomic tests is saliva. Scientists then isolate DNA from the samples and closely examine it, looking for unique patterns in the DNA of the family members who do carry the disease and the DNA of those who don't carry the disease don't have. These unique molecular patterns in the DNA are referred to as polymorphisms, or markers.[9]
遺伝子地図の作成の第一歩は、遺伝子マーカーとマッピング集団の開発である。 2つのマーカーが染色体上で近ければ近いほど、それらが一緒に次世代に受け継がれる可能性が高くなる。 したがって、すべてのマーカーの「共分離」パターンを使用して、それらの順序を再構築することができる。 この点を念頭に置いて、各遺伝子マーカーの遺伝子型を両親と次世代の各個体について記録する。遺伝子地図の質は、地図上の遺伝子マーカーの数とマッピング集団の規模という要因に大きく依存する。この2つの要因は相互に関連しており、マッピング集団が大きければ地図の「解像度」が向上し、「飽和」を防ぐことができる。
The first steps of building a genetic map are the development of genetic markers and a mapping population. The closer two markers are on the chromosome, the more likely they are to be passed on to the next generation together. Therefore, the "co-segregation" patterns of all markers can be used to reconstruct their order. With this in mind, the genotypes of each genetic marker are recorded for both parents and each individual in the following generations. The quality of the genetic maps is largely dependent upon these factors: the number of genetic markers on the map and the size of the mapping population. The two factors are interlinked, as a larger mapping population could increase the "resolution" of the map and prevent the map from being "saturated".
遺伝子マッピングでは、2つの親から正確に区別できる配列の特徴はすべて遺伝マーカーとして使用できる。この点において、遺伝子は2つの親の間で正確に区別できる「形質」によって表される。他の遺伝マーカーとの連鎖は、それらが共通マーカーであるかのように計算され、実際の遺伝子座は、2つの最も近い近隣マーカー間の領域に括弧で囲まれる。その後、特定の原因遺伝子座が特定されるまで、その領域を標的とするより多くのマーカーを調べ、遺伝子の近傍をより高い解像度でマッピングするプロセスが繰り返される。このプロセスは「位置クローニング」と呼ばれることが多く、植物種の研究では広く用いられている。位置クローニングが特に活用されている植物種としては、トウモロコシがある。[10] 遺伝子マッピングの大きな利点は、表現型の影響のみに基づいて遺伝子の相対的位置を特定できることである。
In gene mapping, any sequence feature that can be faithfully distinguished from the two parents can be used as a genetic marker. Genes, in this regard, are represented by "traits" that can be faithfully distinguished between two parents. Their linkage with other genetic markers is calculated in the same way as if they are common markers and the actual gene loci are then bracketed in a region between the two nearest neighboring markers. The entire process is then repeated by looking at more markers that target that region to map the gene neighborhood to a higher resolution until a specific causative locus can be identified. This process is often referred to as "positional cloning", and it is used extensively in the study of plant species. One plant species, in particular in which positional cloning is utilized is in maize.[10] The great advantage of genetic mapping is that it can identify the relative position of genes based solely on their phenotypic effect.
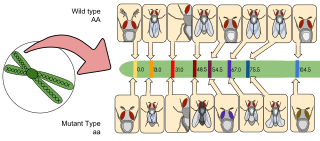
遺伝子マッピングは、どの染色体にどの遺伝子が存在するかを正確に特定し、その遺伝子が特定の染色体上のどの位置にあるかを正確に特定する方法である。マッピングはまた、2つの遺伝子の間の距離に基づいて、どの遺伝子が最も組み換えられやすいかを決定する方法としても機能する。2つの遺伝子の間の距離は、センチモルガンまたはマップユニットと呼ばれる単位で測定される。これらの用語は交換可能である。1つの遺伝子から生じる100個の減数分裂の1つが組み換えを起こす場合、その遺伝子間の距離は1センチモルガンである。[11][12] 2つの遺伝子が互いに離れているほど、それらが組み換えを起こす可能性は高くなる。逆に、2つの遺伝子が近ければ、その逆が起こる。[13]
Genetic mapping is a way to identify exactly which chromosome has which gene and exactly pinpointing where that gene lies on that particular chromosome. Mapping also acts as a method in determining which gene is most likely to recombine based on the distance between two genes. The distance between two genes is measured in units known as centimorgan or map units, these terms are interchangeable. A centimorgan is a distance between genes for which one product of meiosis in one hundred is recombinant.[11][12] The farther two genes are from each other, the more likely they are going to recombine. If it were closer, the opposite would occur.[13]
Linkage analysis
連鎖解析の基礎となるのは、染色体の位置を理解し、疾患遺伝子を特定することである。遺伝的に互いに関連している、または互いに連鎖している特定の遺伝子は、同じ染色体上に近接して存在する。減数分裂の際、これらの遺伝子は一緒に遺伝することが可能であり、疾患の表現型を特定する手助けとなる遺伝マーカーとして使用することができる。連鎖解析は遺伝パターンを特定できるため、これらの研究は通常、家族を対象として行われる。[14]
The basis to linkage analysis is understanding chromosomal location and identifying disease genes. Certain genes that are genetically linked or associated with each other reside close to each other on the same chromosome. During meiosis, these genes are capable of being inherited together and can be used as a genetic marker to help identify the phenotype of diseases. Because linkage analysis can identify inheritance patterns, these studies are usually family based.[14]


初期の遺伝子地図は、トーマス・ハン・モーガンを中心とする研究グループがショウジョウバエの連鎖解析によって作成した。最初のものは1913年に発表された。[16]
The earliest gene maps were done by linkage analysis of fruitflies, in the research group around Thomas Hunt Morgan. The first was published in 1913.[16]
Gene association analysis
遺伝子関連解析は集団ベースであり、遺伝パターンに焦点を当てるのではなく、むしろ集団の全歴史に基づく。遺伝子関連解析では特定の集団を調査し、罹患した個人の対立遺伝子の頻度が、同じ集団の罹患していない対照群の対立遺伝子の頻度と異なるかどうかを特定しようとする。この方法はメンデル遺伝パターンを持たない複雑な疾患を特定するのに特に有用である。[17]
Gene association analysis is population based; it is not focused on inheritance patterns, but rather is based on the entire history of a population. Gene association analysis looks at a particular population and tries to identify whether the frequency of an allele in affected individuals is different from that of a control set of unaffected individuals of the same population. This method is particularly useful to identify complex diseases that do not have a Mendelian inheritance pattern.[17]
Physical mapping
実際の塩基対の距離は一般的に直接測定が困難であるか、不可能であるため、物理地図は、まずゲノムを階層的に小さな断片に分割することによって作成される。各断片を特徴づけ、再び組み立てることで、これらの小さな断片の重複する経路、すなわち「タイル化経路」から、研究者はゲノムの特徴間の物理的距離を推測することができる。
Since actual base-pair distances are generally hard or not possible to directly measure, physical maps are actually constructed by first shattering the genome into hierarchically smaller pieces. By characterizing each single piece and assembling back together, the overlapping path or "tiling path" of these small fragments would allow researchers to infer physical distances between genomic features.
制限酵素地図法は、制限酵素を用いてDNAのセグメントに関する構造情報を取得する方法である。制限酵素は、DNAの特定の認識配列でDNAのセグメントを切断するのを助ける酵素である。制限酵素地図法の基本は、制限酵素でDNAを消化(切断)することである。消化されたDNA断片は、電気泳動法を用いてアガロースゲル上で電気泳動される。これにより、消化された断片のサイズに関する情報が得られる。これらの断片のサイズは、分析されたDNA上の制限酵素部位間の距離を示すのに役立ち、分析されたDNAの構造に関する情報を研究者に提供する。[17] 結果として得られるDNAの移動パターン、すなわちその遺伝子フィンガープリントは、クローン内のどのDNA領域かを特定するために使用される。フィンガープリントを分析することで、コンティグは自動(FPC)または手動(パスファインダー)で重複するDNA領域に組み立てられる。これにより、研究対象の生物のDNA配列を決定するために、クローンを効率的に配列決定するための適切な選択が可能となる。
Restriction mapping is a method in which structural information regarding a segment of DNA is obtained using restriction enzymes. Restriction enzymes are enzymes that help cut segments of DNA at specific recognition sequences. The basis to restriction mapping involves digesting (or cutting) DNA with restriction enzymes. The digested DNA fragments are then run on an agarose gel using electrophoresis, which provides one with information regarding the size of these digested fragments. The sizes of these fragments help indicate the distance between restriction enzyme sites on the DNA analyzed, and provides researchers with information regarding the structure of DNA analyzed.[17] The resulting pattern of DNA migration – its genetic fingerprint is used to identify what stretch of DNA is in the clone. By analyzing the fingerprints, contigs are assembled by automated (FPC) or manual means (pathfinders) into overlapping DNA stretches. Now a good choice of clones can be made to efficiently sequence the clones to determine the DNA sequence of the organism under study.
物理マッピングでは、マッピングには形質や機能に関する情報が含まれないため、特定の遺伝子を直接マーキングする方法はない。遺伝マーカーは、in situ ハイブリダイゼーションのようなプロセスによって物理地図にリンクさせることができる。このアプローチにより、物理地図のコンティグを遺伝地図に「固定」することができる。次に、物理地図のコンティグで使用されたクローンをローカルな規模で配列決定し、新しい遺伝マーカーの設計と原因遺伝子座の特定に役立てる。
In physical mapping, there are no direct ways of marking up a specific gene since the mapping does not include any information that concerns traits and functions. Genetic markers can be linked to a physical map by processes like in situ hybridization. By this approach, physical map contigs can be "anchored" onto a genetic map. The clones used in the physical map contigs can then be sequenced on a local scale to help new genetic marker design and identification of the causative loci.
マクロ制限は、制限酵素の数が少ない制限部位を持つ高分子量DNAを制限酵素で消化する物理的マッピングの一種である。
Macrorestriction is a type of physical mapping wherein the high molecular weight DNA is digested with a restriction enzyme having a low number of restriction sites.
クローンの完全な配列決定を行わずに、クローン群のDNAの重複部分を決定する方法もある。マップが決定されれば、クローンはゲノムの大きな領域を効率的に含むリソースとして使用できる。この種のマッピングは遺伝子マップよりも正確である。
There are alternative ways to determine how DNA in a group of clones overlaps without completely sequencing the clones. Once the map is determined, the clones can be used as a resource to efficiently contain large stretches of the genome. This type of mapping is more accurate than genetic maps.
Restriction mapping
Restriction mapping is a method in which structural information regarding a segment of DNA is obtained using restriction enzymes. Restriction enzymes are enzymes that help cut segments of DNA at specific recognition sequences. The basis to restriction mapping involves digesting (or cutting) DNA with restriction enzymes. The digested DNA fragments are then run on an agarose gel using electrophoresis, which provides one with information regarding the size of these digested fragments. The sizes of these fragments help indicate the distance between restriction enzyme sites on the DNA analyzed, and provides researchers with information regarding the structure of DNA analyzed.[17]
Fluorescent in situ hybridization
制限酵素地図法は、制限酵素を用いてDNAのセグメントに関する構造情報を取得する方法である。制限酵素は、DNAの特定の認識配列でDNAのセグメントを切断するのを助ける酵素である。制限マッピングの基本は、制限酵素でDNAを消化(切断)することである。消化されたDNA断片は、電気泳動法によりアガロースゲル上で電気泳動され、これにより、これらの消化された断片のサイズに関する情報が得られる。これらの断片のサイズは、分析されたDNA上の制限酵素部位間の距離を示すのに役立ち、分析されたDNAの構造に関する情報を研究者に提供する。[17]
Fluorescence in situ hybridization (FISH) is a method used to detect the presence (or absence) of a DNA sequence within a cell.[18] DNA probes that are specific for chromosomal regions or genes of interest are labeled with fluorochromes. By attaching fluorochromes to probes, researchers are able to visualize multiple DNA sequences simultaneously. When a probe comes into contact with DNA on a specific chromosome, hybridization will occur. Consequently, information regarding the location of that sequence of DNA will be attained. FISH analyzes single stranded DNA (ssDNA). Once the DNA is in its single stranded state, the DNA can bind to its specific probe.[6]
Sequence-tagged site (STS) mapping
シーケンスタグ付きサイト(STS)は、個人のゲノム内に複数回出現することが確認されている、短いDNA配列(100~500塩基対の長さ)である。これらのサイトは容易に識別でき、通常、分析対象のDNA内に少なくとも1回は出現する。これらのサイトには通常、遺伝的多型が含まれているため、有効な遺伝子マーカーのソースとなる(他の配列とは異なるため)。シーケンスタグ付きサイトは、ゲノム内でマッピングすることができ、重複するDNA断片のグループを必要とする。一般的に、DNA断片の集合体を作成するためにPCRが使用される。重複する断片が作成された後、STS間のマップ距離を分析することができる。STS間のマップ距離を計算するために、研究者は2つのマーカー間の切断が起こる頻度を決定する(ショットガンシーケンスを参照)[17]。
A sequence-tagged site (STS) is a short sequence of DNA (about 100 - 500 base pairs in length) that is seen to appear multiple times within an individual's genome. These sites are easily recognizable, usually appearing at least once in the DNA being analyzed. These sites usually contain genetic polymorphisms making them sources of viable genetic markers (as they differ from other sequences). Sequenced tagged sites can be mapped within our genome and require a group of overlapping DNA fragments. PCR is generally used to produce the collection of DNA fragments. After overlapping fragments are created, the map distance between STSs can be analyzed. In order to calculate the map distance between STSs, researchers determine the frequency at which breaks between the two markers occur (see shotgun sequencing)[17]
Mapping mutational sites
1950年代初頭には、染色体中の遺伝子はそれぞれ独立した存在であり、遺伝子組み換えによって分割することはできず、連なったビーズのように配置されているという見方が一般的であった。1955年から1959年にかけて、ベンザーはバクテリオファージT4のrII突然変異体を用いて遺伝子組み換え実験を行った。彼は、組み換え実験の結果、突然変異の部位は直線状にマッピングできることを発見した。[19][20] この結果は、遺伝子がDNAの長さに相当する直線構造を持ち、独立して突然変異を起こすことのできる多くの部位があるという主要な考え方を裏付ける証拠となった。
In the early 1950s the prevailing view was that the genes in a chromosome are discrete entities, indivisible by genetic recombination and arranged like beads on a string. During 1955 to 1959, Benzer performed genetic recombination experiments using rII mutants of bacteriophage T4. He found that, on the basis of recombination tests, the sites of mutation could be mapped in a linear order.[19][20] This result provided evidence for the key idea that the gene has a linear structure equivalent to a length of DNA with many sites that can independently mutate.
1961年、フランシス・クリック、レスリー・バーネット、シドニー・ブレナー、リチャード・ワッツ=トビンは、タンパク質の遺伝暗号の基本的な性質を示す遺伝子実験を行った。[21] バクテリオファージT4のrIIB遺伝子内の突然変異部位のマッピングを含むこれらの実験により、遺伝子のDNAの3つの連続した核酸塩基が、コード化されたタンパク質の各アミノ酸を指定することが示された。したがって、遺伝暗号は3つの塩基の組み合わせであり、その3つの組み合わせ(コドンと呼ばれる)が特定のアミノ酸を指定していることが示された。また、タンパク質をコードするDNA配列において、コドンが互いに重複することはないこと、また、そのような配列は固定された開始点から読み取られるという証拠も得られた。
In 1961, Francis Crick, Leslie Barnett, Sydney Brenner and Richard Watts-Tobin performed genetic experiments that demonstrated the basic nature of the genetic code for proteins.[21] These experiments, involving mapping of mutational sites within the rIIB gene of bacteriophage T4, demonstrated that three sequential nucleobases of the gene's DNA specify each successive amino acid of its encoded protein. Thus the genetic code was shown to be a triplet code, where each triplet (called a codon) specifies a particular amino acid. They also obtained evidence that the codons do not overlap with each other in the DNA sequence encoding a protein, and that such a sequence is read from a fixed starting point.
エドガーら[22]は、バクテリオファージT4のr変異体を用いたマッピング実験を行い、rII変異体間の組み換え頻度は厳密に相加的ではないことを示した。2つのrII変異体の交配(a x d)による組み換え頻度は、通常、隣接する内部サブ区間(a x b)+(b x c)+(c x d)の組み換え頻度の合計よりも少ない。厳密に足し合わせることはできないが、系統的な関係が示され[23]、これは遺伝子組み換えの根本的な分子メカニズムを反映している可能性が高い。
Edgar et al.[22] performed mapping experiments with r mutants of bacteriophage T4 showing that recombination frequencies between rII mutants are not strictly additive. The recombination frequency from a cross of two rII mutants (a x d) is usually less than the sum of recombination frequencies for adjacent internal sub-intervals (a x b) + (b x c) + (c x d). Although not strictly additive, a systematic relationship was demonstrated[23] that likely reflects the underlying molecular mechanism of genetic recombination.
Genome sequencing
ゲノムシーケンスは、生物学者ではない人々によって「ゲノムマッピング」と誤って呼ばれることがある。ショットガンシークエンシング[24]のプロセスは、物理マッピングのプロセスに似ている。ゲノムを小さな断片に粉砕し、各断片を特徴づけ、その後それらを再びまとめる(より最近のシークエンシング技術は大幅に異なる)。範囲、目的、プロセスがまったく異なるが、ゲノムアセンブリは、従来の物理マッピングが提供できるすべての情報をはるかに優れた方法で提供するという点で、物理マップの「究極」の形と見なすことができる。
Genome sequencing is sometimes mistakenly referred to as "genome mapping" by non-biologists. The process of shotgun sequencing[24] resembles the process of physical mapping: it shatters the genome into small fragments, characterizes each fragment, then puts them back together (more recent sequencing technologies are drastically different). While the scope, purpose and process are totally different, a genome assembly can be viewed as the "ultimate" form of physical map, in that it provides in a much better way all the information that a traditional physical map can offer.
遺伝子の特定は通常、ある種のゲノムを理解する最初のステップである。遺伝子のマッピングは通常、遺伝子の特定の最初のステップである。遺伝子マッピングは通常、多くの重要な下流の研究の出発点である。
Identification of genes is usually the first step in understanding a genome of a species; mapping of the gene is usually the first step of identification of the gene. Gene mapping is usually the starting point of many important downstream studies.
Disease association
疾患の原因となる遺伝的要素を特定するプロセスは、「マッピング」とも呼ばれる。検索対象となる遺伝子座がすでにかなり限定されている場合、その検索は遺伝子のファインマッピングと呼ばれる。この情報は、大家族における疾患の症状の調査(遺伝的連鎖)や、集団を対象とした遺伝的関連研究から得られる。
The process to identify a genetic element that is responsible for a disease is also referred to as "mapping". If the locus in which the search is performed is already considerably constrained, the search is called the fine mapping of a gene. This information is derived from the investigation of disease manifestations in large families (genetic linkage) or from populations-based genetic association studies.
上述の方法を用いることで、研究者は疾患遺伝子をマッピングすることが可能となる。遺伝子地図の作成は、疾患遺伝子を特定するための重要な第一歩である。遺伝子地図により、変異対立遺伝子を特定でき、変異表現型を引き起こしていると考えられる遺伝子について予測を立てることができる。連鎖解析により特定された疾患の例としては、嚢胞性線維症がある。例えば、嚢胞性線維症(CF)では、CFに罹患した50家族のDNAサンプルが連鎖解析により分析された。CFに関連する数百のマーカーがゲノム全体で分析され、CFが第7染色体長腕に特定されるまでになった。その後、研究者は第7染色体内のさらなるDNAマーカーの連鎖分析を完了し、CF遺伝子のより正確な位置を特定した。CF遺伝子は7q31-q32付近に存在することが判明した(染色体命名法を参照)。[17]
Using the methods mentioned above, researchers are capable of mapping disease genes. Generating a gene map is the critical first step towards identifying disease genes. Gene maps allow for variant alleles to be identified and allow for researchers to make predictions about the genes they think are causing the mutant phenotype. An example of a disorder that was identified by Linkage analysis is Cystic Fibrosis. For example, with Cystic Fibrosis (CF), DNA samples from fifty families affected by CF were analyzed using linkage analysis. Hundreds of markers pertaining to CF were analyzed throughout the genome until CF was identified on the long arm of chromosome 7. Researchers then had completed linkage analysis on additional DNA markers within chromosome 7 to identify an even more precise location of the CF gene. They found that the CF gene resides around 7q31-q32 (see chromosomal nomenclature).[17]
- Eukaryotic chromosome fine structure
- Fate mapping
- G banding
- Genetic fingerprinting
- Genome project
- Human Genome Project
- Optical mapping
- Quantitative trait locus
- Sulston score
- Genomes 3. New York, NY: Garland Science Publishing. (2007). ISBN 9780815341383. OCLC 444522997
- Genomes 3. New York, NY: Garland Science Publishing. (2007). ISBN 9780815341383. OCLC 444522997
Wikiwand in your browser!
Seamless Wikipedia browsing. On steroids.
Every time you click a link to Wikipedia, Wiktionary or Wikiquote in your browser's search results, it will show the modern Wikiwand interface.
Wikiwand extension is a five stars, simple, with minimum permission required to keep your browsing private, safe and transparent.