トップQs
タイムライン
チャット
視点
ポリメラーゼ連鎖反応
DNAサンプルの特定領域を増幅させる反応または技術 ウィキペディアから
Remove ads
ポリメラーゼ連鎖反応(ポリメラーゼれんさはんのう、英語: polymerase chain reaction)とは、DNAサンプルの特定領域を増幅させる反応。

一般的には数百万〜数十億倍に増幅する。英語読みもされるが、その頭文字を取ってPCR法、あるいは単純にPCRと呼ばれることが多い[1][2]。
解説
DNAポリメラーゼと呼ばれる酵素の働きを利用して、一連の温度変化のサイクルを経て任意の遺伝子領域やゲノム領域のコピーを指数関数的に増幅することで、少量のDNAサンプルからその詳細を解析するに十分な量にまで増幅することが目的である[3][4][5]。医療や分子生物学や法医学などの分野で広く使用されている有用な技術である。1983年にキャリー・マリス(Kary Mullis)によって発明され[6][7]、これによりマリスはノーベル賞を受賞した。
PCR法が確立したことにより、DNA配列クローニングや配列決定、遺伝子変異誘導といった実験が容易かつ迅速になった。分子遺伝学や生理学、分類学などの研究分野で活用されている。古代DNAサンプルの解析、法医学や親子鑑定などで利用されるDNA型鑑定、感染性病原体の特定や感染症診断に関わる技術開発(核酸増幅検査)、などにも用いられる。また、PCR法から逆転写ポリメラーゼ連鎖反応やリアルタイムPCR、DNAシークエンシング等の技術が派生して開発されている。今日では、生物学や医学をはじめとする幅広い分野における遺伝子解析の基礎となっている[8][9]。
Remove ads
原理
要約
視点

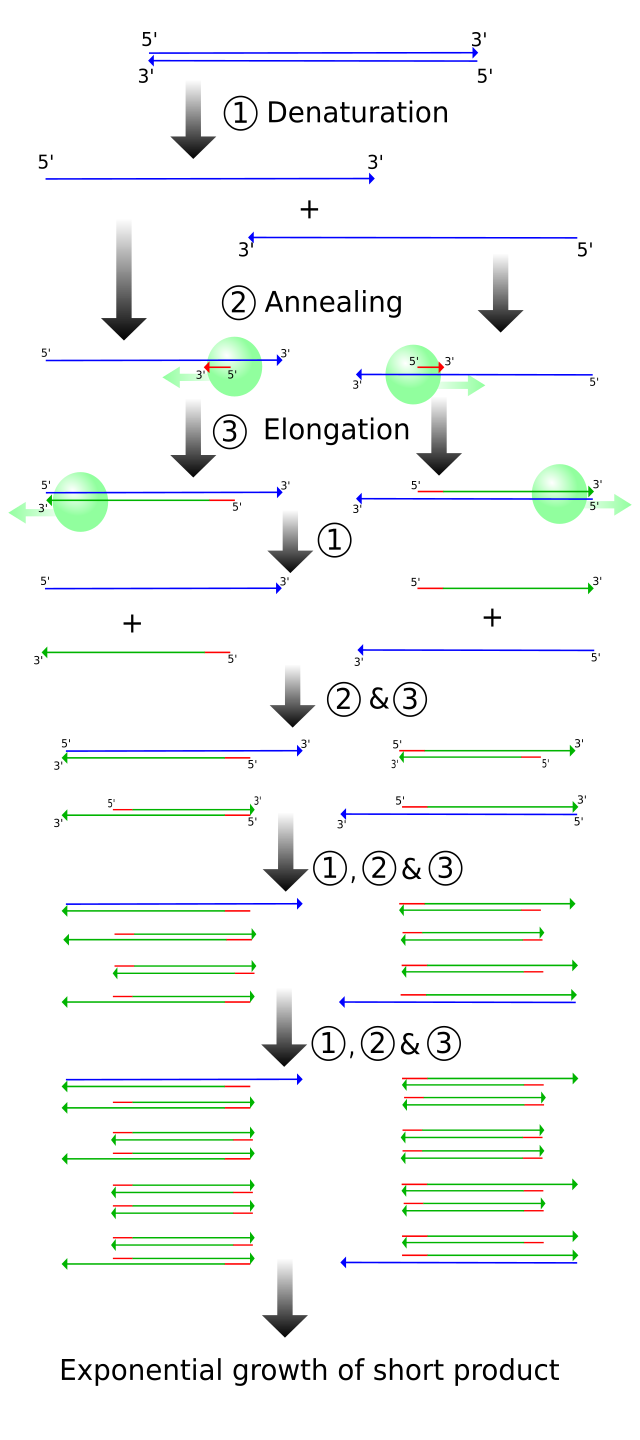
PCR法は、試薬を混交したDNA溶液の温度を上げて下げる、という一連の熱サイクルによって動作する。このDNAサンプルの加熱と冷却の繰り返しサイクルの中で、二本鎖DNAの乖離、プライマーの結合、酵素反応によるDNA合成、という3つの反応が進み、最終的に特定領域のDNA断片が大量に複製される。
PCR法では、増幅対象(テンプレート)のDNAサンプルの他に、大量のプライマー(標的DNA領域に相補的な配列を持つ短い一本鎖DNA〈オリゴヌクレオチド〉)とDNAの構成要素である遊離ヌクレオチド、そしてポリメラーゼの一種であるDNA合成酵素(DNAポリメラーゼ)という3つの試薬を使用する。

- 最初のステップでは、DNA二重らせんの2本鎖DNAを高温下で変性させ、1本鎖DNAに物理的に分離する。変性が起こる温度は、DNAの塩基構成および長さ(塩基数)によって異なり、一般に長いDNAほど温度を高くする必要がある。
- 次に、この1本鎖DNAを含む溶液を冷却して、プライマーを一本鎖DNAの相補配列部位に結合させることで、部分的に2本鎖を作らせる(アニーリング)。冷却が急速であると、長いDNA同士では再結合して2本鎖になることは難しいが、短いDNA断片(オリゴヌクレオチド)は容易に結合できることを利用している。この結果、対象とする長い1本鎖DNAの一部にプライマーが結合したものができる。プライマーをDNAよりも圧倒的に多い状況にしておくことで、DNAとプライマーが結合する傾向はDNAとDNAが結合する傾向よりも、さらに優越的になる。
- 次に、溶液を若干加熱して、この2本鎖DNA部位をテンプレートとしてDNAポリメラーゼを働かせることで、プライマーが結合した部分を起点として、遊離ヌクレオチドを利用して1本鎖部分と相補的なDNAが酵素的に合成される。DNAが合成された後、再び高温にして最初のステップに戻り、このサイクルを最初のDNA変性から繰り返すことにより増幅をすすめる。PCR反応が進むことで、生成されたDNA自体が複製のテンプレートとして使用され、元のDNAテンプレートが指数関数的に増幅される連鎖反応が進む。
以上のようにPCR法は、DNA鎖長による変性とアニーリングの進行速度の違いを利用して、反応溶液の温度の上下を繰り返すだけでDNA合成を繰り返し、任意のDNAの部分領域を増幅する技術である。
使用するDNAポリメラーゼが熱に弱い場合、変性ステップの高温下でDNAとともにポリメラーゼも変性してしまい、失活してしまう。そのためPCR法の開発当初は、DNA変性時の毎回にDNAポリメラーゼを酵素として追加しており、手間と費用がかかっていた[10]。現在では、サーマスアクアティカスという好熱菌由来の熱安定性DNAポリメラーゼであるTaqポリメラーゼなどを用いることで、途中で酵素の追加をせずに反応を連続して進めることができる。
Remove ads
手順
要約
視点
準備
増幅対象のDNA領域の両端の塩基配列を決定し、対応するプライマーを人為合成する。このときプライマーは、増幅予定の2本鎖DNAの両鎖それぞれの3'側に結合する相補配列であり、通常20塩基程度である。多くの場合、実験室でカスタムメイドされるか、商業的な生化学サプライヤーから購入可能である。
反応液調製
増幅対象DNA、プライマー、DNAポリメラーゼおよびDNA合成の素材(基質)であるデオキシヌクレオチド三リン酸(dNTP)、そして酵素が働く至適塩濃度環境をつくるためのバッファー溶液を混合し、PCR装置(サーマルサイクラー)にセットする。流通しているPCR試薬キットに付属するバッファー溶液には二価カチオンが含まれていることが多い[11]。通常はマグネシウムイオン (Mg2+)であるが、PCR媒介DNA突然変異誘発が高いマンガンイオン(Mn2+)を使用することで、あえてDNA合成の間のエラー率を増加させることも可能である[12]。Taq DNAポリメラーゼの場合、一価カチオンとしてカリウムイオン(K+)が入れられることもある[13]。また場合によっては硫酸アンモニウムを加えることもある。アンモニウムイオン(NH4+)は特にミスマッチなプライマーとテンプレート塩基対間の弱い水素結合を不安定化する効果があり、特異性を高めることができる[13]。
PCRサイクル
- 反応液を94°C程度に加熱し、30秒から1分間温度を保ち、2本鎖DNAを1本鎖に分かれさせる(図①)。
- 60°C程度(プライマーによって若干異なる[注 1])にまで急速冷却し、その1本鎖DNAとプライマーをアニーリングさせる(図②)。
- プライマーの分離がおきずDNAポリメラーゼの活性に至適な温度帯まで、再び加熱する。実験目的により、その温度は60–72°C程度に設定される。DNAが合成されるのに必要な時間、増幅する長さによるが通常1〜2分、この温度を保つ(図③)。
- ここまでが1つのサイクルで、以後、①から③までの手順を繰り返していく事で特定のDNA断片を増幅させる。
PCR処理をn回のサイクルを行うと、1つの2本鎖DNAから目的部分を2n-2n倍に増幅する。ただし、通常は最高で35サイクル程度行なう事から、近似的には2nの項は無視できる大きさになる。サイクル数をさらに増やすと、時間経過によりDNAポリメラーゼが活性を失い、またdNTPやプライマーなどの試薬が消費し尽くされるため、反応が制限されて最終的には一連の反応は停止する。
留意点
この反応の成否は、増幅対象DNAとプライマーの塩基配列、サイクル中の各設定温度・時間などに依存する。それらが不適切な場合、無関係なDNA配列を増幅したり、増幅が見られないことがある。また、合成過程において変異が起こる可能性も少なからずあるため、使用目的によっては生成物の塩基配列のチェックが必要である。
PCRの応用
要約
視点
DNAの増幅と定量
PCRはターゲットのDNAの領域を劇的に増幅する技術であり、非常に少量のDNAサンプルであっても、PCRを経ることで分析を可能になる場合がある。このことは、証拠として極微量のDNAしか入手できない法医学などの分野においては、特に重要である。あるいは、例えば数万年前の古代のDNAの分析などにもPCRは威力を発揮する[14]。
定量PCR(リアルタイムPCR、あるいは単純にqPCRとも呼ばれる。RT-PCRとは異なることに注意)の技術確立により、サンプル中に存在する特定のDNA配列の量も推定することができる[15]。これは、遺伝子発現レベルを定量的に決定する用途などで利用されている。定量的PCRでは、PCRサイクルのプロセスを実行中に、PCRサイクルの中で増幅されてゆくPCR産物の濃度をリアルタイムで測定していくことで、元々存在したターゲットのDNA領域の存在量を定量化することができる。大きく2つの手法があり、一つは二本鎖の間に非特異的に保持される蛍光色素を使用する方法、もう一つは予め蛍光標識が付加され特定の配列をコードしたプローブを利用した方法である。後者の方法では、プローブとその相補DNAのハイブリダイゼーションが行われることで初めて蛍光を検出することができる。
リアルタイムPCRと逆転写反応を組み合わせたRT-qPCR(逆転写ポリメラーゼ連鎖反応)と呼ばれる手法では、DNAではなくRNAの定量を可能にしている。この技術では、まず最初にmRNAをcDNAに変換し、そのcDNAをqPCRによって定量化する。この手法は、がんなどの遺伝病に関連する遺伝子の検出や発現量測定に多く利用されている[16]。
生物学研究への応用
PCRは分子生物学や遺伝学をはじめとする、様々な研究分野に応用されている。
- PCRを用いてゲノム中の特定のDNA領域を選択的に増幅し分離することができる。このようなPCRの利用は、サザンブロッティングやノーザンブロッティングといったハイブリダイゼーション用プローブの生成や、特定のDNA領域に由来した大量のDNA断片を必要とするDNAクローニングなどで広く行われている。
- PCRはDNAシーケンスを行う上でもしばしば重要である。様々なPCRにより、例えば完全に未知のゲノムから解析対象の遺伝子配列やDNA領域を抽出して増幅したりできる。
- PCRは、DNAクローニングなどの古典的な実験プロセスで多く活用されている。例えば大きなゲノムから、特定のゲノム領域をベクターに挿入する際などに利用される。また、すでにベクターに挿入されているDNA断片を分析したり増幅するために利用することもできる。PCRプロトコルを一部変更することで、挿入断片の突然変異を人為的に誘発することもできる。
- 古代DNAを対象とした研究にも、PCRはよく活用される。このような古代DNAは大部分が紫外線や加水分解により分解されており、極微量の二本鎖DNAしか存在しない場合が多いため、PCRで増幅をかけることではじめて解析を行うことが可能になる。実際にPCRを用いた研究例としては、ネアンデルタール人の骨、4万年前のマンモスの凍結組織、エジプトのミイラの脳などがあり、他にはロシア皇帝やイギリス王リチャード3世の同定などが行われている[14]。場合によっては、かなりの程度分解されてしまったDNAサンプルであっても、PCR増幅をかけることである程度元のDNA配列を復元することができる可能性がある。
- 遺伝子発現のパターンの研究にもPCRが利用される。体組織や個々の細胞をさまざまな時系列段階で分析して、どの遺伝子が活性化/非活性化したのかを調べることができるほか、定量PCRを使用して実際の発現レベルを詳細に定量化することもできる。
- PCRは遺伝的連鎖の研究にも利用されている。例えば、個々の精子からいくつかの遺伝子座を同時に増幅し、減数分裂後の染色体クロスオーバーを調べた研究が報告されている[17]。この研究では、数千の精子を分析することで、非常に近い遺伝子座間のまれなクロスオーバーイベントが直接観察されている。同様に、異常な欠失、挿入、転座、または反転を分析することができる。
- PCRを使用して、任意の遺伝子やゲノム領域の部位特異的な突然変異を誘発することができる。これらの変異体を調べることで、例えばタンパク質の機能を解明したり、あるいはタンパク質の機能を変更または改善する研究を進めることができる。
出生前診断
PCRは、子供が生まれる前に特定の遺伝の保因者であるかどうか、あるいは実際に病気に冒されているかどうかをテストする、いわゆる出生前診断に利用することができる[18]。出生前検査用のDNAサンプルは、羊水穿刺による絨毛膜絨毛サンプリング、あるいは母親の血流中を循環するごく少量の胎児細胞の分析によって取得できる。PCR分析は着床前診断も不可欠であり、発生中の胚の個々の細胞の突然変異をテストできる。
臓器移植の組織タイピング
PCRは、臓器移植に不可欠な組織タイピングの高感度な試験としても使用できる。血液型に対する従来の抗体ベースのテストをPCRベースのテストに置き換える提案も、2008年にされている[19]。
がんの遺伝子分析
がんの多くの形態は、がん発生に関連する各種遺伝子(がん遺伝子)の配列変化を伴うが、PCR技術を活用してこの突然変異を分析することで、治療方針を患者に合わせて個別にカスタマイズできる可能性がある。またPCRは、白血病やリンパ腫などの悪性疾患の早期診断を可能にする。がん研究分野で開発が進められており、現在ではすでにPCRは日常的に使用されている。ゲノムDNAサンプルを直接PCRでアッセイすることで、転座特異的悪性細胞を他の方法よりも少なくとも10,000倍高い感度で検出できることが報告されている[20]。PCRはまた、腫瘍抑制因子の分離と増幅も可能にする。たとえば、定量PCRを使用して単一細胞を定量し、DNAやmRNA、タンパク質の存在量と組み合わせを解析することが可能である[21]。
感染症の診断
PCRは、細菌やウイルスによって引き起こされる感染症の、高感度で迅速な診断に役立っている[22]。PCRでは、マイコバクテリア や嫌気性細菌、または組織培養アッセイや動物モデルからのウイルスなど、培養できない微生物や成長の遅い微生物の迅速な同定も可能である。またPCR診断は、感染性病原体の検出のみならず、その細菌が特定の遺伝子を持っているかどうかを判断することで、非病原性株か病原性株かを区別できる[22][23]。一方で、様々な欠点も報告されている(後述)。
- ヒト免疫不全ウイルス (HIV)は、発見して根絶するのが難しい標的である。初期の感染診断は、血流中を循環するウイルス抗体の存在に依存していたが、抗体は感染後何週間も経ってからでないと現れず、母体の抗体は新生児の感染を隠してしまい、またHIV治療薬による治療では抗体量が変化しないという問題があった。そこで、50,000以上の細胞DNAサンプル中からわずか1つのウイルスゲノムを検出できる高感度PCRテストの手法が開発された[24]。この方法により、感染症の早期検出や献血された血液のウイルス検査、新生児の迅速な感染検査か可能になり、また抗ウイルス治療の効果を定量化できるようになった。
- 結核のようないくつかの病気を引き起こす微生物は、患者からのサンプリングが難しく、また実験室で成長するのが遅いことが知られており、培養ベースの診断では多くの手間暇がかかっていた。PCRによるテストにより、サンプル中から疾患原因微生物を検出できるほか、遺伝子分析から抗生物質耐性の有無等も検出でき、効果的な治療方針の設定や治療効果の評価に繋がる可能性がある。
- 家畜または野生動物の集団を介した疾患生物の拡散や新しい毒性を持つサブタイプの出現は、PCRテストによって監視できる。
- ウイルスのDNAの標的配列に特異的なプライマーを使用することで、ウイルスDNAをPCRで検出することができるほか、DNAシーケンスにも使用できる。PCRの感度が高いため、感染直後および病気の発症前であってもウイルス検出が可能な場合がある[25]。早期発見により、医師に治療の重要なリードタイムを与える可能性がある。患者の内部に含まれるウイルス量は、PCRベースのDNA定量技術によっても定量化できる。
- 百日咳は百日咳菌と呼ばれる細菌によって引き起こされる。この細菌は、さまざまな動物や人間に影響を与える深刻な急性呼吸器感染症を特徴としており、多くの幼い子供の死をもたらしている。百日咳毒素は、2つの二量体によって細胞受容体に結合し、細胞免疫に役割を果たすTリンパ球などと反応する、タンパク質毒素である[26]。PCRにより百日咳毒素遺伝子内にある配列を検出できるため、培養法と比較して非常に効率的に百日咳の診断が可能である[27]。
法医学への応用
PCRベースのDNA型鑑定(フィンガープリンティング)は、法医学分野で広く応用されている。
- DNA型鑑定は、世界中の全人口から一人を一意に区別することができる技術である。この技術を応用し、微量のDNAサンプルを犯罪現場から採取して分析し、囚人のDNAデータベースから比較することで、容疑者を特定できる場合がある。より簡易な利用方法としては、犯罪捜査中に容疑者を迅速に除外するために利用される。あるいはまた、数十年前もの前の犯罪証拠品をテストすることで、有罪判決を受けた人々の犯行を確認したり、あるいは免罪することにも繋がる。
- 法医学におけるDNAタイピング(フォレンジックDNAタイピング)は、犯罪現場で発見された証拠の分析から犯罪容疑者を特定または除外する効果的な方法である。ヒトゲノムには、遺伝子配列内またはゲノムの非コード領域に見られる多くの反復領域がある。具体的には、ヒトDNAの最大40%が反復的であることが知られている[28]。ゲノム内のこれらの反復非コード領域には2種類あり、一つは可変長タンデムリピート(VNTR)と呼ばれ、10〜100塩基対の長さである一方で、他方はショートタンデムリピート(STR)と呼ばれ、2〜10塩基対の繰り返しセクションで構成されます。そして、各反復領域に隣接するプライマーを使用してPCR増幅をかけることができる。各個人から取得したいくつかのSTRのフラグメントのサイズの分布を調べると、統計的に高い確率で、各個人を一意に特定することができる[28]。さらに、ヒトゲノムの完全配列はすでに決定されており、この配列情報はNCBI Webサイトから簡単にアクセスできるため、様々な応用がなされている。たとえばFBIでは識別に使用するDNAマーカーサイトのセットを編集しており、これらは結合DNAインデックスシステム(CODIS)DNAデータベースと呼ばれている[28]。このデータベースを使用すると、DNAサンプルが一致する確率を統計的に決定できる。分析に使用するターゲットDNAの量が非常に少なくて済むため、PCRはフォレンジックDNAタイピングに使用する非常に強力で重要な分析ツールである。たとえば、毛包が付着した人間の髪には、1本もあれば分析を行うのに十分なDNAが含まれている。同様に、数個の精子、指の爪の下からの皮膚サンプル、または少量の血液は、決定的な分析に十分なDNAを提供できる[28]。
- 一方で逆に、識別力の低い形式によるDNAフィンガープリンティングは、親子鑑定に活用されている。この場合、身元不明の人間の遺体といった対象者は予想される近親者、すなわち親や兄弟、子供等のDNAと比較される。養子になった(誘拐された)子供の生物学的な両親を確認するためや、新生児の実際の生物学的父親の確認 (または除外)などにも利用される。
- アメロゲニン遺伝子を対象としたPCRでは、法医学的な骨サンプルからリアルタイムで男女の性決定をする事ができる。これにより、古代標本や犯罪容疑者の性別を判別することができる[29]。
Remove ads
PCRの技術的制約
PCRには原理と実際の作業は非常に簡単で、結果を迅速に得ることができ、また非常に感度が高い、といった多くの利点がある。定量PCR (qPCR、quantitative PCR) ではさらに、ターゲットとなったDNA領域の定量化もできる利点がある。一方で、PCRには様々な技術的制約や限界も知られている。
PCRの技術的な制限の1つに、選択的増幅を可能にするプライマーを生成するために、ターゲット領域の配列に関する事前情報が必要なことが挙げられる[30]。すなわち、PCR実施者は通常、プライマーとテンプレートが適切に結合するように、事前にターゲットとなるDNA領域の前後の配列情報を知っておく必要がある。そのため、配列情報が完全に未知のターゲットに対しては、PCRをかけることは原則的に不可能である。また、他のあらゆる酵素と同様であるが、DNAポリメラーゼ自体もDNA合成時にエラーを起こしやすく、生成されるPCR増幅物の配列に変異が生じることがある[31]。さらに、PCRはごく少量のDNAでも増幅できるため、誤って混入したDNAを元に増幅が起きてしまい、曖昧な結果や誤った結果が生じることがある。
このような問題を回避し、PCR条件を最適化するため、多くの手法と手順が開発されている[32][33]。例えば、サンプルが外来DNAの混入によって汚染されてしまう可能性を最小限に抑えるために、試薬の準備とPCR処理・分析、の各ステップで別々の部屋を利用することで、両者を空間的に分離することが有効である[34]。また、サンプルや試薬の操作には常に使い捨ての新品チューブ類やフィルター付きピペットチップを使用し、作業台や機器は徹底的に洗浄して常にきれいな空間で作業することが有効である[35]。PCR産物の収量を改善して偽産物の形成を回避する上でプライマー設計を見直すこと、バッファーやポリメラーゼ酵素の種類を検討することも、また重要である。バッファーシステムにホルムアミドなどの試薬を添加すると、PCRの特異性と収量が増加する場合がある[36]。プライマー設計を支援するための、理論的なPCR結果のコンピューターシミュレーション (Electronic PCR) も開発されている[37]。
Remove ads
感染症診断におけるPCRの特徴
要約
視点
PCRは非常に強力で実用的な研究ツールであり、実際に多くの感染症において、病因の配列決定はPCRを用いて解明されている。この手法は、既知ウイルスや未知ウイルスの識別に役立ち、疾患自体の理解に大きく貢献している。手順をさらに簡素化でき、高感度の検出システムを開発できれば、PCRは今後臨床検査室の重要な位置を占めるようになると考えられている[38]。しかしながら、感染症診断におけるPCRの利用には、利点のみならず様々な欠点も指摘されている。
利点
- ヒトのゲノム(30億塩基対)のような長大なDNA分子を含む検体中から、特定のDNA断片(数百から数千塩基対)だけを選択的に増幅させることができる[3]。
- 極めて微量なDNA量で目的を達成できる。
- 短時間で増幅反応を完了できる[4]。増幅に要する時間が2時間以下程度と短い。
- 使用機器(PCR機)等は共通のまま、病原体ごとに特異的なプライマーを用いることで検査が可能である[4]。
- 病原体は死滅していても(感染力を失っていても)標的核酸が保存されていれば増幅でき、危険な病原体でも不活化してから検査することができる[4]。
- Nested PCR、リアルタイムPCRなどを利用することで感度を上げることができる[4]。
欠点
- 臓器や組織など生体材料を検体とする場合、採材部位や材料の前処理によって検出率が異なり、結果が陰性であっても生体における病原体の存在は必ずしも否定できない[4]。
- 核酸の増幅には反応阻害物の生成などによる反応効率の低下などの影響があるため限界があり、核酸が検出可能な量にまで増幅できなければ結果は陰性となるが、そのような場合には病原体の存在を否定できない[4]。
- 血液・糞便など生体材料の検査では検査材料中の阻害物質の影響を受けることがあるため精製作業が必要とされるが、それでも完全ではないとされている[4]。また、検査材料の保存状態や凍結融解の回数、核酸精製の方法・条件などが検査効率や検査結果に大きな影響を与えることがある[4]。
- 検査時の陽性対照からの汚染、以前の検査や実験に由来する汚染、試薬類への核酸の混入が誤った陽性を招く危険性がある(コンタミネーション)[4]。
歴史と背景
Kjell Kleppeとハー・ゴビンド・コラナらは、プライマーと短いDNAテンプレートを使用して酵素アッセイをin vitroで行う手法を、1971年にJournal of Molecular Biology(分子生物学ジャーナル)に最初に発表した[39]。これはPCRの基本的な原理を説明したものであったが、当時あまり注目されておらず、ポリメラーゼ連鎖反応の発明は一般的に1983年のキャリー・マリスの功績によるものとみなされている[40]。
1983年にマリスがPCRを開発したとき、彼はカリフォルニア州エメリービルで、最初のバイオテクノロジー企業の1つであるシータス社(Cetus Corporation)で働いていた。マリスは「ある夜、Pacific Coast Highwayを車でドライブ中に、PCRのアイデアを思いついた」と書いている[41]。彼は、DNAの変化(突然変異)を分析する新しい方法を考えていた時、当時すでに知られていたオリゴヌクレオチドとDNAポリメラーゼを用いたDNA合成反応を繰り返すことで核酸の部分領域を増幅することを思いついた[41]。
マリスはこの方法を "polymerase-catalyzed chain reaction"(ポリメラーゼ触媒連鎖反応)と名付け、ネイチャーやサイエンスなどの著名な科学雑誌に論文として投稿したが、掲載されなかった。一方、PCR法自体はシータス社の同僚の手により鎌状赤血球症という遺伝性疾患の迅速な診断手段に応用された。サイエンス誌に "Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia"として報告され、オリジナル論文より前に世界の科学者の注目を集めることとなった[7]。1987年にようやく、マリスの論文は Methods in Enzymology 誌に"Specific synthesis of DNA in vitro via a polymerase-catalyzed chain reaction."として掲載された[42]。後にマリスはサイエンティフィック・アメリカンで、「PCRは、遺伝物質DNAの単一分子から始めて、午後には1,000億の類似した分子を生成できる。反応は簡単に実行できる。試験管、いくつかの簡単な試薬、および熱源を必要とするだけである」と記述している[43]。DNAフィンガープリンティングは1988年に父子鑑定に初めて使用された[44]。
この成果を評価され、マリスはシータス社の同僚と共に、PCR技術を立証してから7年後の1993年にノーベル化学賞を受賞した[45]。また、1985年のR.K. SaikiおよびH.A. Erlichによる“Enzymatic Amplification of β-globin Genomic Sequences and Restriction Site Analysis for Diagnosis of Sickle Cell Anemia”(「鎌状赤血球貧血の診断のためのβグロビンゲノムシーケンスの酵素的増幅および制限部位分析」)の論文が、2017年の米国化学会の化学史部門の化学ブレイクスルー賞を受賞した[46][47]。しかしながら、マリスの研究に対する他の科学者の貢献や、彼がPCR原理の唯一の発明者であったかどうかに関しては、以下に記述するように、いくつかの論争が残っている。
PCRは当初、大腸菌のDNAポリメラーゼIをズブチリシン処理し、5'-3'エキソヌクレアーゼ活性を除去したクレノー断片を用いて反応を起こすものが大半であった。しかしながらこの酵素は、各複製サイクル後のDNA二重らせんの分離に必要な高温に耐えられず、DNAポリメラーゼが失活してしまうために、サーマルサイクルごとに手作業でこの酵素を加える必要があった[48]。そのため、DNA複製の初期手順は非常に非効率的で時間がかかり、プロセス全体で大量のDNAポリメラーゼと継続的な処理が必要であった。シータス社の研究グループは、この欠点を解決するために、50〜80°Cもの高温環境(温泉)に住んでいる好熱性細菌であるサーマス・アクアティクス(T. aquaticus)[49]から、耐熱性DNAポリメラーゼとしてTaqポリメラーゼを精製し、これを用いたPCRの手法を1976年にサイエンス誌に発表した[8]。T. aquaticusから単離されたDNAポリメラーゼは、90 °C (194 °F)超える高温で安定であり、DNA変性後も活性を維持する[50]ため、各サイクル後に新しいDNAポリメラーゼを追加する必要がなくなる[51]。これにより、PCR反応の簡便化と自動化への道が開かれ、幅広く応用可能な手法として発展することになった。
このように、PCR法の応用、発展に関してはシータス社グループ(当初はマリスも含む)の果たした役割が大きいのである。
ただし最初にこの方法を着想し方向性を示したのはキャリー・マリスであるので、マリスがノーベル化学賞を1993年に受賞した。PCR技術はマリスが特許を取得し、1983年にマリスが技術を発明したときに働いていたシータス社に譲渡された。Taqポリメラーゼ酵素も特許で保護されている。デュポンが提起した不成功の訴訟を含む、この技術に関連するいくつかの有名な訴訟が存在した。スイスの製薬会社エフ・ホフマン・ラ・ロシュは、1992年に特許権を購入したが、現在その特許権は失効している[52]。
Remove ads
PCRの種類・応用法
- Conventional PCR
- 1組のプライマーで反応を25〜35サイクル程度繰り返す通常のPCR[4]。
- Real-time polymerase chain reaction(Real-Time PCR、リアルタイムPCR)
- 核酸断片を発光させて専用の光学機器を使って検出することによってリアルタイムに増幅曲線を描く方法。定性ではなく定量が必要な時に用いる。[4]。
- Reverse transcription polymerase chain reaction(RT-PCR、逆転写ポリメラーゼ連鎖反応)
- 逆転写酵素によりRNAをcDNAにしてからPCRを行う方法[4]。
- Nested polymerase chain reaction(Nested PCR)
- PCRで増幅したPCR産物を改めて次の反応のテンプレートにして、テンプレート内部に設計した別のプライマーペアを用いてもう一度PCRを繰り返す方法[4]。
- Multiplex polymerase chain reaction(Multiplex PCR、マルチプレックスPCR)
- 複数の標的核酸(DNA)のそれぞれのPCR反応を1本の反応チューブ内で同時に行う方法[4]。
- Amplified fragment length polymorphism(AFLP)
- 標的核酸(DNA)を含むゲノムDNA等を制限酵素で切断し、その切断末端に短い2本鎖DNA(アダプター)を結合させ、その相補的なプライマーを用いてPCRを行う方法[4]。
- Loop-Mediated Isothermal Amplification(LAMP法)
- 従来のPCRとは異なる原理に基づく方法[4][53]。
Remove ads
脚注
関連項目
Wikiwand - on
Seamless Wikipedia browsing. On steroids.
Remove ads
