Nucleosoma
unidades básicas por medio das que se realiza a compactación do ADN nos eucariotas From Wikipedia, the free encyclopedia
Remove ads
Os nucleosomas son as unidades básicas por medio das que se realiza a compactación do ADN nos eucariotas. Constan dun segmento de ADN enrolado arredor dun núcleo de proteínas histonas.[1] Esta estrutura é a miúdo comparada a un fío enrolado nun carrete.[2]
Os nucleosomas forman as unidades repetidas básicas da cromatina eucariótica,[3] que se usan para empaquetar os grandes xenomas eucarióticos no núcleo permitindo ao mesmo tempo un adecuado acceso a el (en células de mamíferos debe empaquetarse aproximadamente unha lonxitude de 2 m de ADN dentro dun núcleo duns 10 µm de diámetro). A cadea de nucleosomas pode pregarse formando unha serie de estruturas de orde progresivamente superior, ata orixinar finalmente os cromosomas; isto compacta o ADN e á vez crea un novo nivel de control regulatorio que asegura a correcta expresión dos xenes. Pénsase que os nucleosomas levan información epixenética herdada en forma de modificacións covalentes no seu núcleo de histonas. Don e Ada Olins [4] e Roger D. Kornberg.[5][6] propuxeron en 1974 a hipótese do nucleosoma para explicar isto.
O núcleo do nucleosoma consta de aproximadamente 147[7] pares de bases de ADN enrolado á esquerda dando 1,67 voltas superhelicoidais arredor do octámero de histonas formado por dúas copias de cada unha das catro histonas: H2A, H2B, H3, e H4.[8] As partículas do núcleo (core) do nucleosoma están conectadas por tramos de "ADN espazador", que pode ter uns 80 pares de bases de longo. Tecnicamente, o nucleosoma defínese como dito núcleo máis unha desas rexións espazadoras de ADN; aínda que a palabra se usa con frecuencia como sinónimo do núcleo ou core.[9]
As histonas de unión (linker) como a H1 e as súas isoformas están implicadas na compactación da cromatina e sitúanse na base do nucleosoma preto dos puntos de entrada e saída do ADN e unidas ás rexións espazadoras do ADN.[10] Os nucleosomas non condensados sen as histonas de unión (H1) semellan vistos ao microscopio electrónico as "doas dun colar de ADN".[11]
A diferenza da maioría das células eucarióticas, as células espermáticas maduras usan en gran medida protaminas para compactar o seu ADN xenómico, moi probablemente para acadar un grao de compactación aínda maior.[12] Nas arqueas atopáronse tamén equivalentes das histonas e unha estrutura da cromatina simplificada,[13] o que proba que os eucariotas non son os únicos organismos que posúen nucleosomas.
Remove ads
Estrutura
Estrutura do núcleo (core) do nucleosoma

Visión de conxunto
Os primeiros estudos estruturais xa forneceron probas de que o ADN rodeaba un octámero de histonas dando case dúas voltas nunha superhélice levoxira. En 1997 O grupo de Richmond resolveu a primeira estrutura cristalina do nucleosoma con resolución case atómica, na que se mostraban algúns dos principais detalles desta partícula. Hai que sinalar, porén, que o o ADN palindrómico alfa-satélite humano, fundamental para conseguir a estrutura cristalina do nucleosoma de 1997, obtívose en realidade polo grupo de Bunick no Laboratorio nacional de Oak Ridge, en Oak Ridge, Tennessee.[14][15] O grupo de investigación de Bunick foi pioneiro no desenvolvemento do ADN palindrómico alfa-satélite humano e isto foi un avance esencial para poder determinar a estrutura do núcleo do nucleosoma con alta resolución.[16][17][18]
Ata agora foron resoltas as estruturas duns 20 núcleos de nucleosomas distintos,[19] incluíndo aqueles que contiñan variantes das histonas normais e histonas de diferentes especies. A estrutura do núcleo ou core do nucleosoma está extraordinariamente conservada na evolución, e medidas comparativas de nucleosomas de especies tan diferentes como o sapo Xenopus e o lévedo Saccharomyces mostran unha desviación estatística mínima.[20]
O núcleo ou core do nucleosoma
A partícula nuclear do nucleosoma (mostrada na figura) consta dunhas 146[7] pares de bases de ADN enrolado dando 1,67 voltas de superhélice levoxira arredor do octámero de histonas, e consta de dúas copias de cada unha das catro histonas core H2A, H2B, H3, e H4. Os nucleosomas contiguos están unidos por un tramo de ADN libre chamado ADN espazador ou linker DNA (que varía en lonxitude de 10 - 80 pares de bases dependendo da especie e tipo de tecido[13]).
Interaccións proteicas no nucleosoma
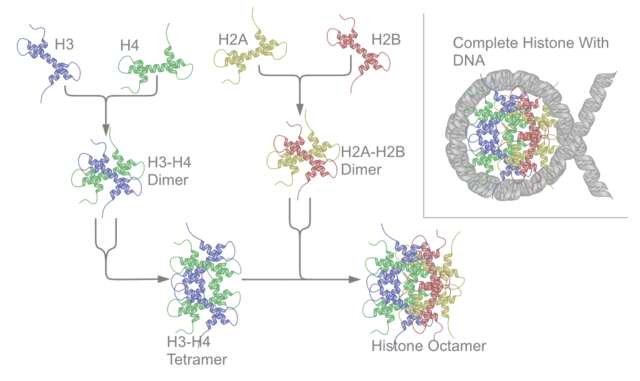
As proteínas histonas do octámero conteñen un motivo estrutural característico denominado "pregamento de histona" que consiste en tres alfa-hélices (α1-3) separadas por dous bucles (L1-2). En disolución as histonas forman heterodímeros H2A-H2B e heterotetrámeros H3-H4. As histonas dimerízanse polas súas longas hélices α2 con orientación antiparalela, e no caso das H3 e H4, dous destes dímeros forman un grupo de 4 hélices estabilizado por unha intensa interacción H3-H3. O dímero H2A/H2B únese ao tetrámero H3/H4 debido a interaccións entre H4 e H2B, as cales inclúen a formación dun agrupamento hidrofóbico.[8] O octámero de histonas está formado por un tetrámero central H3/H4 que queda en medio de dous dímeros H2A/H2B. Debido á forte carga básica (positiva) dos catro tipos de histonas do octámero, o octámero é só estable en presenza do ADN ou en concentracións salinas moi altas.
Interaccións histona-ADN
No nucleosoma establécense unhas 120 interaccións directas proteína-ADN e varios centos de interaccións indirectas mediadas pola auga.[21] As interaccións directas proteína-ADN non se producen regularmente por toda a superficie do octámero senón que máis ben están localizadas en sitios concretos. Isto débese á formación de dous tipos sitios de unión do ADN no octámero; o sitio α1α1 que usa a forma de hélice α1 de dúas histonas adxacentes e o sitio L1L2 formado polos bucles L1 e L2. As principais interaccións co ADN son enlaces salinos (electrostáticos) e enlaces de hidróxeno entre os grupos básicos e hidroxilo das cadeas laterais dos aminoácidos ou os grupos amida da cadea proteínica, e os fosfatos do ADN. Isto é importante dado que a ubicua distribución dos nucleosomas nos xenomas require que o factor de unión ao ADN non sexa específico da secuencia. Aínda que os nucleosomas tenden a preferir algunhas secuencias do ADN máis ca outras,[22] poden unirse a practicamente calquera secuencia, o que se pensa que se debe á flexibilidade na formación desas interaccións mediadas por moléculas de auga. Ademais, establécense interaccións non polares entre cadeas laterais das proteínas e os grupos desoxirribosa, e unha cadea lateral de arxinina intercálase no suco menor do ADN en todos os 14 puntos en que este queda fronte á superficie do octámero. A distribución e a forza dos sitios de unión do ADN sobre a superficie do octámero distorsiona o ADN situado sobre o núcleo do nucleosoma. O ADN non está uniformemente dobrado e contén defectos de torsión. A torsión da forma B do ADN libre en disolución é de 10,5 pares de bases por volta, pero a torsión total do ADN do nucleosoma é de só 10,2 pares de bases por volta, variando desde un valor de 9.4 a 10.9 pares de bases por volta.
Dominios da cola das histonas
As extensión con forma de cola das histonas supoñen o 30% da masa das histonas, pero non son visibles nas estruturas cristalinas do nucleosoma debido á súa grande flexibilidade, e crese que están moi desestructuradas.[23] As colas N-terminais das histonas H3 e H2B pasan a través dunha canle formada polos sucos menores das dúas cadeas do ADN, sobresaíndo do ADN cada 20 pares de bases. Por outra parte, a cola N-terminal da histona H4 ten unha rexión de aminoácidos moi básicos (16-25), a cal, na estrutura cristalina, establece unha interacción coa rexión superficial fortemente acídica do dímero H2A-H2B doutro nucleosoma, o que é potencialmente relevante para establecer estruturas de orde superior nos nucleosomas. Esta interacción pénsase que acontece tamén en condicións fisiolóxicas e suxire que a acetilación da cola da H4 distorsiona as estruturas de orde superior da cromatina.
Estruturas de orde superior

O grao de organización do ADN ao que se chega nos nucleosomas non explica totalmente a compactación do ADN observada no núcleo. Cómpre que exista unha compactación da cromatina de grao superior, pero non se comprende totalmente. Os coñecementos actuais[19] son que a sucesión de nucleosomas co seu ADN espazador forman a fibra de 10 nm da cromatina, coñecida descritivamente como o "colar de doas", que ten unha razón de compactación de aproximadamente 5 a 10.[13] A cadea de nucleosomas pode dispoñerse formando a fibra de 30 nm, unha estrutura máis compacta cunha razón de compactación de ~50[13], cuxa formación depende da presenza da histona H1.
Presentouse unha estrutura cristalina de tetranucleosoma que se usou para elaborar e propoñer unha estrutura en 2-start hélice da fibra de 30 nm.[24] Pero hai aínda bastantes reservas sobre este modelo, xa que é incompatible con datos recentes obtidos con microscopía electrónica.[25] Á parte disto, a estrutura da cromatina non se comprende ben, pero a suxestión clásica é que a fibra de 30 nm se dispón formando bucles ao longo dun armazón proteico central para formar a eucromatina transcricionalmemnte activa. Unha maior compactación orixinaría a heterocromatina transcricionalmente inactiva.
Remove ads
Dinámica dos nucleosomas
Aínda que o nucleosoma é un complexo proteína-ADN moi estable, non é estático e demostrouse que sofre un certo número de cambios estruturais, incluíndo o reposicionamento do nucleosoma e cambios no sitio de exposición de ADN. Dependendo da situación, os nucleosomas poden inhibir ou facilitar a unión de factores de transcrición. As posicións dos nucleosomas están controladas por tres factores principais: Primeiro, a afinidade intrínseca de unión do octámero de histonas depende da secuencia de ADN. Segundo, o nucleosoma pode ser desprazado ou recrutado pola unión competitiva ou cooperativa doutros factores proteicos. Terceiro, o nucleosoma pode ser translocado activamente por complexos remodeladores dependentes do ATP.[26] [27] [28]
Remove ads
Modulación da estrutura do nucleosoma
Os xenomas eucarióticos están sempre asociados á cromatina; non obstante, as células necesitan regular tanto espacial coma temporalmente loci específicos dentro da gran masa de cromatina. Para conseguir o alto grao de control requirido para coordinar procesos nucleares tales como a replicación do ADN, a súa reparación e a transcrición, as células desenvolveron varios xeitos de modular a estrutura e función da cromatina de forma local e específica. Isto pode implicar modificacións covalentes das histonas, incorporación de variantes das histonas normais, e a remodelación non covalente feita por encimas remodeladores dependentes do ATP.[13][29][30][31][32][33]
Ensamblaxe dos nucleosomas in vitro
Os nucleosomas poden ensamblarse in vitro tanto usando histonas nativas purificadas coma histonas recombinantes.[34][35] Unha técnica estándar para enrolar o ADN arredor das histonas require o uso da diálise de sales. Nesta técnica incúbanse xuntos octámeros de histonas e un molde ADN espido a unha concentración salina 2 M. Reducindo a modo a concentración salina, o ADN equilíbrase nunha posición na que se enrola arredor dos octámeros de histonas, formando os nucleosomas. En condicións apropiadas, este proceso de reconstitución permite mapear experimentalmente a afinidade do nucleosoma por posicionarse nunha determinada secuencia.[36]
Remove ads
Notas
Véxase tamén
Wikiwand - on
Seamless Wikipedia browsing. On steroids.
Remove ads