Sitio CpG
From Wikipedia, the free encyclopedia
Remove ads
Os sitios CpG ou sitios CG son rexións do ADN nas que un nucleótido de citosina vai seguido dun nucleótido de guanina na secuencia linear de bases en dirección 5' → 3'. Os sitios CpG aparecen con altas frecuencias en rexións xenómicas chamadas illas CpG (ou illas CG). As citosinas dos dinucleótidos CpG poden ser metiladas para formar 5-metilcitosinas. Os enzimas que engaden estes grupos metilo denomínanse ADN metiltransferases. En mamíferos, do 70% ao 80% das citosinas dos sitios CpG están metiladas.[1] Metilar citosinas nun xene pode cambiar a súa expresión e é un mecanismo que forma parte dun amplo eido da bioloxía que estuda a regulación denominado epixenética.
Remove ads
Características dos CpG
Definición
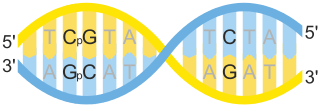
CpG é a abreviación de 5'—C—fosfato—G—3' , é dicir, nucleósidos de citosina e guanina separados só por un grupo fosfato (do enlace fosfodiéster); o fosfato liga cada dous nucleósidos contiguos no ADN (ao ter un fosfato o nucleósido denomínase nucleótido). A notación CpG utilízase para distinguir esta secuencia linear situada nunha mesma febra do ADN dun apareamento de bases CG de citosina e guanina entre secuencias de distintas febras do ADN. A notación CpG debe interpretarse, pois, como que a citosina está en posición 5 prima (5') con respecto á guanina. Non debe confundirse CpG con GpC, xa que este último significa que a guanina está seguida da citosina na dirección 5' → 3' dunha secuencia nunha soa febra do ADN.
Infrarrepresentación no xenoma
Os dinucleótidos CpG aparecen con moita menos frecuencia na secuencia dos xenomas de vertebrados do que sería de esperar por pura probabilidade. Por exemplo, no xenoma humano, que ten un 42% de contido GC,[2] esperaríase que aparecese un par de nucleótidos consistente en citosina seguida de guanina nun 0,21 × 0,21 = 4,41%, pero a frecuencia observada de CpG nos xenomas humanos é de 0,98%, menos da cuarta parte do que se agardaba.[3] Esta infrarrepresentación de CpG é unha consecuencia da alta taxa de mutación que presentan os sitios CpG metilados: mentres que a desaminación espontánea da citosina orixina uracilo, o cal é unha base estraña (no ADN) que é rapidamente substituída por unha citosina polo mecanismo de reparación por escisión de bases; a desaminación dunha citosina metilada orixina unha timina, e as bases G:T resultantes discordantes (o normal é G:C ou A:T) son a miúdo resoltas indebidamente durante a reparación como A:T. A taxa de transcición de C a T nos sitos CpG metilados é ~10 veces maior que nos sitios non metilados.[4][5][6][7]
Distribución xenómica
Os dinucleótidos CpG aparecen frecuentemente nas chamadas illas CpG (ver definición de illa CpG máis abaixo). No xenoma humano hai 28 890 illas CpG, (que son 50 267 se incluínos as illas CpG que aparecen en secuencias repetidas).[8] Isto está de acordo coas 28 519 illas CpG atopadas por Venter et al.,[9] xa que a secuencia xenómica de Venter et al.non inclúe os interiores de elementos repetitivos altamente similares e as rexións repetidas extremadamente densas preto dos centrómeros.[10] Como as illas CpG conteñen múltiples secuencias de dinucleótidos CpG, parece que hai máis de 20 millóns de dinucleótidos CpG no xenoma humano.
Remove ads
Illas CpG

As illas CpG (ou illas CG) son rexións cunha alta frecuencia de sitios CpG. Aínda que as definicións obxectivas de illa CpG son limitadas, a definición formal habitual é que son unha rexión de polo menos 200 pares de bases, unha porcentaxe GC maior do 50% e unha proporción observado-esperado de CpG maior do 60%. A "razón observado-esperado de CpG" pode derivarse cando os observados se calculan como:
e os esperados como:
ou
Moitos xenes nos xenomas de mamíferos teñen illas CpG asociadas co comezo dun xene[13] (as rexións promotoras). Debido a isto, a presenza dunha illa CpG utilízase para axudar á predición e anotación de xenes.
Nos xenomas de mamíferos, as illas CpG son normalmente de entre 300 e 3 000 pares de bases de lonxitude, e atopáronse en aproximadamente o 40% dos promotores dos xenes de mamíferos.[14] Un 70% dos promotores humanos teñen unha alta concentración de CpG. Dada a frecuencia de secuencias de dous nucleótidos GC, o número de dinucleótidos CpG é moito menor do esperado.[12]
Un estudo de 2002 revisou as regras da predición de illas CpG para excluír outras secuencias xenómicas ricas en GC como as repeticións Alu. Baseándose nunha ampla investigación sobre a secuencia completa dos cromosomas humanos 21 e 22, as rexións do ADN maiores de 500 pares de bases son as que máis probablemente son "verdadeiras" illas CpG asociadas coas rexións 5' dos xenes se teñen un contido GC maior do 55% e unha razón observado-esperado de CpG do 65%.[15]
As illas CpG caracterízanse por un contido de dinucleótidos CpG de polo menos o 60% do que se esperaría estatisticamente (~4–6%), mentres que o resto do xenoma ten unha frecuencia de CpG moi inferior (~1%), un fenómeno chamado supresión CG. A diferenza dos sitios CpG na rexión codificante dun xene, na maioría dos exemplos de sitios CpG en illas CpG os promotores non están metilados se son xenes que se expresan. Esta observación levou a especular que a metilación dos sitios CpG no promotor dun xene pode inhibir a expresión xénica. A metilación, xunto coas modificacións de histonas, son fundamentais na impronta xenética.[16] A maioría das diferenzas de metilación entre tecidos ou entre mostras normais e cancerosas de tecidos, aparecen a unha curta distancia das illas CpG (nas chamadas "costas das illas CpG") en vez de nas illas propiamente ditas.[17]
As illas CpG adoitan aparecer en ou preto do sitio de inicio de transcrición dos xenes, especialmente os xenes de mantemento (housekeeping genes), en vertebrados.[12] Unha base C (citosina) seguida inmediatamente dunha base G (guanina), é dicir un CpG, é rara no ADN de vertebrados porque as citosinas en dita disposición adoitan estar metiladas. Esta metilación axuda a distinguir a febra de ADN de nova síntese da febra parental, o que facilita as fases finais da corrección de probas do ADN despois da duplicación. Porén, co tempo as citosinas metiladas tenden a transformase en timinas debido á desaminación espontánea. Existe un enzima especial en humanos, a timina-ADN glicosilase ou TDG, que substitue especificamente as T situadas en discordancias T/G. Porén, debido á rareza dos CpGs, teorízase que non é dabondo efectivo para impedir unha posible mutación rápida dos dinucleótidos. A existencia de illas CpG explícase xeralmente pol existencia de forzas selectivas de contido CpG relativamente alto ou baixos niveis de metilación nesa área xenómica, o que quizais ten que ver coa regulación da expresión xénica. Recentemente, un estudo mostrou que maioría das illas CpG son o resultado de forzas non selectivas.[18]
Remove ads
Metilación, silenciamento, cancro e envellecemento

- Artigo principal: Metilación do ADN.
Illas CpG en promotores
En humanos, un 70% dos promotores localizados preto do sitio de inicio da transcrición dun xene (promotores proximais) conteñen unha illa CpG.[19][20]
Os elementos promotores distais tamén conteñen frecuentemente illas CpG. Un exemplo é o xene para a reparación do ADN ERCC1, no que o elemento que contén a illa CpG está localizado a uns 5 400 nucleótidos augas arriba do sitio de inicio da transcrición do xene ERCC1.[21] As illas CpG tamén aparecen frecuentemente en promoteres para ARNs non codificantes funcionais como os microARNs.[22]
A metilación de illas CpG silencia establemente xenes
Nos humanos a metilación do ADN ocorre na posición 5 do anel de pirimidina dos residuos de citosina dentro dos sitios CpG para formar 5-metilcitosinas. A presenza de sitios CpG metilados múltiples en illas CpG de promotores causa o silenciamento estable de xenes.[23] O silenciamento dun xene pode ser iniciado por outros mecanismos, pero isto adoita ir seguido da metilación dos sitios CpG na illa CpG promotora para causar o silenciamento estable do xene.[23]
Hiper/hipometilación dos CpG do promotor no cancro
En cancros a perda de expresión de xenes ocorre unhas 10 veces máis frecuentemente por hipermetilación de illas CpG do promotor que por mutacións. Como indicaron Vogelstein et al., nun cancro colorrectal adoita haber de 3 a 6 mutacións driver (que lle dan vantaxe ao clon) e de 33 a 66 mutacións autostop ou pasaxeiro (que non dan vantaxe pero están asociadas coas driver).[24] En contraste, nun estudo de tumores de colon comparados con mucosa do colon adxacente aparentemente normal, 1 734 illas CpG estaban fortemente metiladas en tumores, mentes que estas illas CpG non estaban metiladas na mucosa adxacente.[25] A metade das illas CpG estaban en promotores de xenes codificantes de proteínas anotados,[25] o que suxire que uns 867 xenes nun tumor de colon perderon a súa expresión debido á metilación das illas CpG. Un estudo separado encontrou unha media de 1 549 rexións metiladas diferencialmente (hipermetiladas ou hipometiladas) nos xenomas de seis cancros de colon (comparadas coa mucosa adxacente), dos cales 629 estaban en rexións promotoras de xenes coñecidas.[26] Un terceiro estudo atopou máis de 2 000 xenes metilados diferencialmente entre cancros de colon e a mucosa adxacente. Usando a análise de enriquecemento de conxuntos de xenes, 569 dun total de 938 conxuntos de xenes estaban hipermetilados e 369 estaban hipometilados en cancros.[27] A hipometilación de illas CpG en promotores ten como resultado a sobreexpresión dos xenes ou conxuntos de xenes afectados.
Un estudo de 2012[28] elaborou unha lista de 147 xenes específicos con promotores hipermetilados asociados co cancro de colon, xunto coa fecuencia coa cal se atopaban estas hipermetilacións en cancros de colon. Polo menos 10 destes xenes tiñan promotores hipermetilados en case o 100% dos cancros de colon. O estudo tamén indicou 11 microARNs cuxos promotores estaban hipermetilados en cancros de colon con frecuencias entre o 50% e o 100% dos cancros. Os microARNs son pequenos ARNs endóxenos que se aparean con secuencias de ARNs mensaxeiros para dirixir a represión postranscricional. Como media, cada microARN reprime varios centos de xenes diana.[29] Deste xeito, os microARNs con promotores hipermetilados poden permitir a sobreexpresión de centos ou miles de xenes no cancro.
A información anterior mostra que, nos cancros, a hiper/hipometilación de CpG de promotores de xenes e de microARNs causa a perda de expresión (ou ás veces un incremento da expresión) de moitos máis xenes que unha mutación.
Xenes de reparación do ADN con promotores hiper/hipo-metilatdos en cancros
Os xenes de reparación do ADN están frecuentemente reprimidos en cancros debido á hipermetilación de illas CpG dentro dos seus promotores. En carcinomas de célula escamosa de cabeza e pescozo polo menos 15 xenes de reparación do ADN teñen frecuentemente promotores hipermetilados; estes xenes son: XRCC1, MLH3, PMS1, RAD51B, XRCC3, RAD54B, BRCA1, SHFM1, GEN1, FANCE, FAAP20, SPRTN, SETMAR, HUS1, e PER1.[30] Uns dezasete tipos de cancro son frecuentemente deficientes nun ou máis xenes de reparación do ADN debido á hipermetilación dos seus promotores.[31][32] Como exemplo, a hipermetilación do promotor do xene de reparación do ADN MGMT aparece nun 93% dos cancros de vexiga, no 88% dos cancros de estómago, no 74% dos cancros de tiroide, no 40%-90% dos cancros colorrectais e no 50% dos cancros de cerebro. A hipermetilación do promotor de LIG4 ocorre nun 82% dos cancros colorrectais. A hipermetilación do promotor de NEIL1 ocorre no 62% dos cancros de cabeza e pescozo e nun 42% dos cancros pulmonares de células non pequenas. A hipermetilación do promotor de ATM ocorre no 47% dos cancros pulmonares de células non pequenas. A hipermetilación do promotor de MLH1 aprece no 48% dos carcinomas escamosos pulmonares de células non pequenas. A hipermetilación do promotor de FANCB dáse no 46% dos cancros de cabeza e pescozo.
Por outra parte, os promotores de dous xenes: PARP1 e FEN1, estaban hipermetilados e estes xenes sobreexpresábanse en numerosos cancros. O PARP1 e o FEN1 son xenes esenciais na vía de reparación do ADN tendente ao erro chamada unión de extremos mediada por microhomoloxía. Se esta vía se sobreexpresa o exceso de muacións que causa pode orixinar un cancro. O PARP1 sobreexprésase en leucemias activadas por tirosina quinase,[33] no neuroblastoma,[34] en tumores testiculares e noutros de célula xerminal,[35] e no sarcoma de Ewing.[36] O FEN1 está sobreexpresado na maioría dos cancros de mama,[37] próstata,[38] estómago,[39][40] neuroblastomas,[41] cancros pancreáticos,[42] e pulmonares.[43]

Os danos no ADN parecen ser a principal causa subxacente do cancro.[45][46] Se a reparación exacta do ADN é deficiente, os danos no ADN tenden a acumularse. Tal exceso de danos no ADN pode incrementar os erros mutacionais durante a replicación do ADN debido a síntese translesión tendente ao erro. O exceso de danos no ADN pode tamén incrementar as alteracións epixenéticas debido a erros durante a reparación do ADN.[47][48] Ditas mutacións e alteracións epixenéticas poden dar lugar ao cancro (ver neoplasmas malignos). Así, a hiper/hipometilación das illas CpG nos promotores de xenes de reparación do ADN son probablemente fundamentais na progresión do cancro.
Metilación de sitios CpG co envellecemento
Como a idade ten un forte efecto sbre os niveis de metilación do ADN en decenas de miles de sitios CpG, pode definirse un reloxo biolóxico moi exacto (denominado reloxo epixenético ou idade de metilación do ADN) en humanos e chimpancés.[49]
Sitios non metilados
Os dinucleótidos CpG non metilados poden ser detectados polo receptor de tipo Toll 9[50] (TLR 9) en células dendríticas plasmacitoides, monocitos, células asasinas naturais (NK) e linfocitos B en humanos. Isto utilízase para detectar infeccións virais intracelulares.
Remove ads
Os sitios CpG e a memoria

En mamíferos, as ADN metiltransferases (que engaden grupos metilo a bases do ADN) mostran unha preferencia de secuencia polas citosinas que están nos sitios CpG.[52] No cerebro de rato o 4,2% de todas as citosinas están metiladas, principalmente no contexto dos sitios CpG, formando 5mCpG.[53] A maioría dos sitios 5mCpG hipermetilados incrementan a represión de xenes asociados.[53]
Como sinalaron Duke et al., a metilación de ADN de neuronas (que reprime a expresión de xenes particulares) é alterada pola actividade neuronal. A metilación do ADN de neuronas é necesaria para a plasticidade sináptica; é modificada polas experiencias; e cómpre a metilación e desmetilación do ADN activo para a formación e mantemento da memoria.[54]
En 2016 Halder et al.[55] utilizando ratos, e en 2017 Duke et al.[54] usando ratas, someteron os roedores a medo condicionado contextual, causando a formación dunha memoria a longo prazo especialmente forte. Observaron que 24 horas despois do condicionamento, na rexión cerebral do hipocampo das ratas, a expresión de 1 048 xenes estaba regulada á baixa (usualmente asociada coa presenza de 5mCpG nos promotores dos xenes) e a expresión de 564 xenes estaba regulada á alza (a miúdo asociada coa hipometilación de sitios CpG en promotores de xenes). Comprobaron que 24 horas despois do adestramento, o 9,2% dos xenes no xenoma de neuronas do hipocampo de ratas estaban metilados diferencialmente. Porén, aínda que o hipocampo é esencial para a aprendizaxe de nova información non pode almacenar a información. Nos experimentos con ratos de Halder, observáronse no hipocampo 1 206 xenes metilados diferencialmente unha hora despois do condicionamento do medo contextual, mais estas metilacións alteradas foron revertidas e xa non se vían despois de catro semanas. En contraste coa ausencia de cambios na metilación CpG a longo prazo no hipocampo, unha metilación CpG diferencial substancial podía detectarse nas neuronas corticais durante o mantemento da memoria. Había 1 223 xenes metilados diferencialmente no córtex cingulado anterior de ratos durante as catro semanas posteriores ao condicionamento do medo contextual.
A desmetilación en sitios CpG require a actividade de especies reactivas do oxíxeno
Dúas revisións[56][57] resumen a gran cantidade de probas sobre o papel esencial das especies reactivas do oxíxeno (ROS) na formación da memoria. A desmetilación do ADN en miles de sitios CpG durante a formación da memoria depende da súa iniciación por especies reactivas do oxíxeno. En 2016, Zhou et al.,[44] mostraron que as especies reactivas do oxíxeno teñen un papel central na desmetilación do ADN.
A TET1 é un enzima implicado na desmetilación de 5mCpG. Porén, a TET1 só pode actuar sobre a 5mCpG sempre e cando unha especie reactiva do oxíxeno actuase primeiro sobre a guanina para formar 8-hidroxi-2'-desoxiguanosina (8-OHdG), orixinando un dinucleótido 5mCp-8-OHdG (ver a penúltima figura).[44] Despois da formación de 5mCp-8-OHdG, o enzima de reparación por escisión de bases OGG1 úneses á lesión 8-OHdG sen escisión inmediata. A adherencia de OGG1 ao sitio 5mCp-8-OHdG recruta a TET1, permitindo que esta oxide a 5mC adxacente á 8-OHdG, como se ve na penúltima figura. Isto inicia a vía de desmetilación mostrada na última figura.
A expresión de proteínas alterada en neuronas, controlada pola desmetilación dependente de especies reactivas do oxíxeno de sitios CpG en promotores de xenes en ADN de neuronas, é básica para a formación da memoria.[58]
Remove ads
Notas
Wikiwand - on
Seamless Wikipedia browsing. On steroids.
Remove ads





