Loading AI tools
Als Virus-Taxonomie bezeichnet man die international verbindliche Benennung von Viren, Virusfamilien und -gattungen. Die Entscheidung über verschiedene Taxa wird von einem internationalen Gremium, dem International Committee on Taxonomy of Viruses (ICTV), beraten und getroffen.
Die Virus-Taxonomie ist ein an die Taxonomien von Pflanzen und Tieren angelehnter Versuch, die Vielfalt viraler und subviraler Entitäten (auch Prionen und Retrotransposons) systematisch und einheitlich zu ordnen. Sie darf nicht mit der weiter gefassten Virusklassifikation verwechselt werden, bei der verschiedene Eigenschaften von Viren zur Einteilung herangezogen werden können (zum Beispiel nur Genomstruktur, Baltimore-Klassifikation, Wirtsspecies, Übertragungsart etc.).
Vorgänger der heutigen ICTV-Taxonomie ist die von André Lwoff, Robert W. Horne und Paul Tournier im Jahre 1962 entsprechend der von Carl von Linné begründeten binären Klassifikation der Lebewesen begründete System (nach diesen Autoren LHT-System genannt). In ihr werden analog zur Taxonomie anderer Lebewesen, die folgenden Taxa unterteilt:
- Virosphäre (Phylum: Vira)
Die entscheidenden Charakteristika für diese Klassifikation waren:
- die Natur des viralen Genoms (DNA oder RNA)
- die Symmetrie des Kapsids
- Vorhandensein einer Lipidumhüllung
- Größe von Virion und Kapsid
Auf Grundlage des Wissens um die Molekularbiologie der Viren hatte sich ab 1971 eine weitere Klassifikation etabliert, welche auf einen Vorschlag des Nobelpreisträgers David Baltimore zurückgeht.[1] Wichtige Kriterien dieser Baltimore-Klassifikation sind:
- die Genomstruktur:
- die Form (Symmetrie) des Kapsids
- das Vorhandensein einer Hülle
- die Anordnung der Gene innerhalb des Genoms
- die Replikationsstrategie (abhängig von Genomstruktur)
- die Virusgröße
Das International Committee on Taxonomy of Viruses (ICTV) begann Anfang der 1970er Jahre mit der Ausarbeitung und Umsetzung von Regeln für die Benennung und Klassifizierung von Viren, eine Arbeit, die bis in die Gegenwart andauert. Das ICTV ist das einzige Gremium, das von der International Union of Microbiological Societies mit der Aufgabe betraut wurde, eine universelle Virustaxonomie zu entwickeln, zu verfeinern und aufrechtzuerhalten.[2] Das System hat viele Gemeinsamkeiten mit dem Klassifikationssystem der zellulären Organismen, wie z. B. die Taxonstruktur. Es gibt jedoch einige Unterschiede, wie etwa die fest vorgeschriebenen Namensendungen für taxonomische Namen aller Ränge ab Gattung aufwärts, sowie die durchgängige Verwendung von Kursivschrift für alle taxonomischen Namen[3] – anders als für zelluläre Organismen (nach International Code of Nomenclature für Algen, Pilze und Pflanzen und International Code of Zoological Nomenclature).[4]
Gegenüber den Baltimore-Kriterien zielt dabei der Fokus zunehmend auf Übereinstimmungen in der Nukleotidsequenz (Homologie). Dies erlaubt es im Prinzip auch Metagenomanalysen mit einzubeziehen, so dass man für die Aufstellung einer Taxonomie nicht mehr notwendig auf die Isolation eines Virusstamms angewiesen ist. Möglich machen dies die Fortschritte in Vereinfachung, Zugänglichkeit, Dauer und Kosten der Gensequenzierung. Auch wenn die Baltimore-Kriterien an zweiter Stelle immer noch eine wichtige Rolle spielen, geraten sie immer weiter in den Hintergrund. Spezies, über die das ICTV keine ausreichenden Genom-Informationen bekommt, sind daher taxonomisch nicht einzuordnen und werden vom ICTV ggf. auch wieder ausgelistet. Zudem hat das ICTV immer mehr Taxa höheren Ranges aufgestellt, die aufgrund von Gen-Homologien Vertreter verschiedener Baltimore-Gruppen in sich vereinen (z. B. Pleolipoviridae, Ortervirales und zwei der vier Realm – en. realms), und die daher als Gruppe nicht im Baltimore-System unterzubringen sind.
Für die Taxonomie unberücksichtigt, aber dennoch für die Zusammenfassung unterschiedlicher Viren mit gemeinsamen medizinischen oder epidemiologischen Merkmalen wichtige Kriterien sind:
- gemeinsame Organismen (Wirte), die sie infizieren
- ähnliche Krankheitsbilder bzw. Infektion des gleichen Organs (z. B. Hepatitis-Viren)
- gemeinsame Übertragungswege, z. B. Übertragung durch Vektoren (Gliederfüßer: Arboviren)
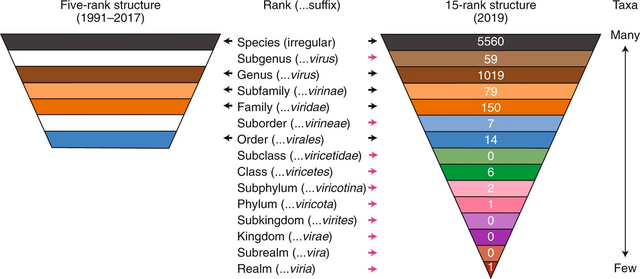
Die taxonomische Struktur war bis 2017 im Prinzip wie bei der herkömmlichen Virusklassifikation ab Stufe Ordnung und darunter (siehe oben) und wurde 2018 durch weitere Stufen wie folgt ergänzt (mit vom LHC-System abweichenden Namensendungen). Oberste Rangstufe ist jetzt Realm[5] (en. realm, anstelle der Domäne bei zellulären Organismen):[6][7]
- Realm (en. realm) (…viria)
- Subrealm (en. subrealm) (…vira) (Endung wie bei Subphylum im LHC-System, als zweiteoberste Stufe)
Für spezielle Fälle sind ausnahmsweise auch bestimmte Abweichungen der Namensform möglich (s. u.).
Es gibt in diesen Richtlinien (bisher) keine Definition von Tribus, Unterarten (Subspezies), Stämmen (analog zu Bakterienstamm, en. strain) oder Isolaten. Die Namen einzelner Viren (im Sinn von Isolaten oder Stämmen) werden – genauso wie reine Akronyme normal (nicht kursiv) geschrieben.[3]
Unter den Viren gibt es zurzeit (Stand 21. Juni 2024, ratifiziert April 2024) folgende Anzahl von Vertretern der jeweiligen taxonomischen Rangstufen:[8]
| Rang | Anzahl Mitglieder |
|---|---|
| Art | 14690 |
| Untergattung | 84 |
| Gattung | 3522 |
| Unterfamilie | 200 |
| Familie | 314 |
| Unterordnung | 11 |
| Ordnung | 81 |
| Unterklasse | 0 |
| Klasse | 41 |
| Subphylum | 2 |
| Phylum | 18 |
| Unterreich | 0 |
| Reich | 10 |
| Subrealm | 0 |
| Realm | 6 |
Virologen haben bei Indischen Riesenflughunden mittels PCR-Techniken nach bekannten und unbekannten Vertretern von neun verschiedenen Virenfamilien respektive -gattungen gesucht, und aufgrund dieser Resultate abgeschätzt, dass die gesamte Säugetierwelt mindestens 320.000 Virenarten beherbergen muss.[9] In der offiziellen Veröffentlichung des ICTV (Stand März 2020, Master Species List 2018b.v2)[10] finden sich aufgrund des starken Zuwachses an Wissen viele sonst übliche Bezeichnungen nicht, da über deren taxonomische Zuordnung bisher noch keine einheitliche Meinung gefunden werden konnte.
Stand: 5. Mai 2024 (MSL#39v2)[11]
Realm (englisch realm ‚Bereich‘)
- Adnaviria (Baltimore 1)[A. 1]
- Duplodnaviria (Baltimore 1)
- Monodnaviria (Baltimore 1 und 2)
- Riboviria (Baltimore 3 bis 7)[A. 2]
- Ribozyviria (Baltimore 5)[A. 3]
- Varidnaviria (Baltimore 1)[A. 4]
Klasse(n) ohne Zuweisung zu einem höheren Rang
- Naldaviricetes (Arthropoda-spezifische Viren mit großem DNA-Genom – Familien Baculoviridae, Hytrosaviridae, Nimaviridae, Nudiviridae)
Familien ohne Zuweisung zu einem höheren Rang
- Alphasatellitidae
- Ampullaviridae
- Anelloviridae
- Avsunviroidae
- Bartogtaviriformidae
- Bicaudaviridae
- Brachygtaviriformidae
- Clavaviridae
- Fuselloviridae
- Globuloviridae
- Guttaviridae
- Halspiviridae
- Itzamnaviridae
- Ovaliviridae
- Plasmaviridae
- Polydnaviriformidae
- Portogloboviridae
- Pospiviroidae
- Rhodogtaviriformidae
- Spiraviridae
- Thaspiviridae
- Tolecusatellitidae
Gattungen ohne Zuweisung zu einem höheren Rang
Im Gegensatz zu den zellulären Organismen (Lebewesen) bilden die Viren in ihrer Gesamtheit keine monophyletische Gruppe, sondern mehrere Verwandtschaftsgruppen mit vermutet unterschiedlicher Entstehungsgeschichte (auch wenn zwischen all diesen Gruppen und den Lebewesen horizontaler Gentransfer möglich ist). Die Realms können als solche maximalen Verwandtschaftsgruppen angesehen werden, bei den Top-Level-Taxa niedrigerer Ränge ist dagegen eine künftige Zuweisung zu neuen oder zu bestehenden Realms möglich (und in der Vergangenheit auch schon geschehen). Eine Virus-Taxonomie, die auch vorgeschlagene Taxa umfasst findet sich beim National Center for Biotechnology Information (NCBI)[14]
Die Viren mit RNA-Genom bilden insgesamt keine gemeinsame Verwandtschaftsgruppe (Klade) und sind daher keine taxonomische Einheit (Taxon). Fast alle RNA-Viren gehören jedoch zum Realm Riboviria, das zusätzlich auch revers transkribierende Viren enthält und die umfassendste Verwandtschaftsgruppe von Viren darstellt. Eine kleinere Gruppe von RNA-Viren gehört zum Realm Ribozyviria; daneben gibt es derzeit (April 2023) noch einige Taxa von RNA-Viren ohne Zuordnung zu einem Realm.
Viren mit RNA-Genom wurden 2018 in ihrer Gesamtheit zunächst geschlossen in den Realm Riboviria gestellt,[15] dieser Realm umfasste damit komplett die (ihrerseits nicht-taxonomischen) Baltimore-Gruppen 3, 4 und 5. Da die Riboviria dadurch charakterisiert sind, dass sie eine eigene RNA-abhängige Polymerase kodieren, wurden im Jahr 2019 die Gruppen wieder ausgegliedert, bei denen das nicht der Fall ist, und teilweise dem neuen Realm Ribozyviria zugeführt. Die revers transkribierenden Viren wurden umgekehrt den Riboviria zugeordnet, da sie solche Polymerasen kodieren.[10][16]
Die Viren mit DNA-Genom bilden ebenfalls keine gemeinsame Verwandtschaftsgruppe (Klade) und daher keine taxonomische Einheit (Taxon). Die Realms von DNA-Viren sind: Adnaviria, Duplodnaviria, Monodnaviria und Varidnaviria, daneben gibt es derzeit (April 2023) noch einige Taxa von DNA-Viren in ohne Zuordnung zu einem Realm.
Die Klassifizierung nach den Baltimore-Kriterien identifiziert nicht immer vollständige Verwandtschaftsgruppen (Kladen). Baltimore-Gruppen umfassen oft mehrere Realms, d. h. Bereiche von Viren vermutlich unterschiedlichen Ursprungs. Umgekehrt erstrecken sich manche Virentaxa über mehrere Baltimore-Gruppen, weil etwa dsDNA-Vertreter und ssDNA-Vertreter offenbar naher verwandt sind. Zu Details der Baltimore-Klassifizierung siehe Virusklassifikation §Die Baltimore-Klassifikation.
Dazu gehören Viren mit positiver Einzelstrang-RNA, die per Reverse Transkriptase (RT) in DNA zurückgeschrieben und ins Zellgenom eingebaut wird (Retroviren (+)ssRNA-RT, Baltimore-Gruppe 6), sowie Viren mit Doppelstrang-DNA, die umgekehrt zur Replikation einen RNA-Zwischenschritt benutzen und daher ebenfalls revers transkribieren (Pararetroviren dsDNA-RT, Baltimore-Gruppe 7). Die meisten dieser Vertreter gehören unabhängig davon der Ordnung Ortervirales an.[17]
- zum Realm Riboviria
- Reich Pararnavirae
- Phylum Artverviricota
- Klasse Revtraviricetes
- Ordnung Blubervirales (dsDNA-RT: Revers transkribierende DNA-Viren)
- Familie Hepadnaviridae (mit Gattung Orthohepadnavirus und darin Spezies Hepatitis-B-Virus)
- Ordnung Ortervirales (bis auf Caulimoviridae revers transkribierende RNA-Viren ssRNA-RT)
- Familie Belpaoviridae (ssRNA-RT)
- Familie Metaviridae (ssRNA-RT)
- Familie Pseudoviridae (ssRNA-RT)
- Familie Retroviridae (ssRNA-RT, mit Gattung Lentivirus und darin den Spezies HIV-1 und -2, SIV, BIV, FIV)
- Familie Caulimoviridae (dsDNA-RT: Revers transkribierende DNA-Viren)
- Ordnung Blubervirales (dsDNA-RT: Revers transkribierende DNA-Viren)
- Klasse Revtraviricetes
- Phylum Artverviricota
- Reich Pararnavirae
Viriforme
Das ICTV hat im März/April 2022 für die bis dato als Viren bezeichneten Vertreter der Familie Polydnaviridae den neuen Begriff „viriform“ eingeführt, den diese Taxa fortan im Namen tragen. Insbesondere ist die neue Bezeichnung dieser Gruppe nun
- Familie Polydnaviriformidae
- Gattung: Bracoviriform
- Gattung: Ichnoviriform
Weitere Familien Viriformer sind (Stand 30. April 2024):[11]
- Familie Bartogtaviriformidae
- Gattung Bartonegtaviriform
- Familie Brachygtaviriformidae
- Gattung Brachyspigtaviriform
- Familie Rhodogtaviriformidae
- Gattung Dinogtaviriform
- Gattung Rhodobactegtaviriform
- Gattung Rhodovulugtaviriform
- Gattung Ruegerigtaviriform
Viroide
Die Taxonomie für Viroide nach ICTV folgt denselben Regeln wie für Viren, nur dass der Namensbestandteil „viroid“ enthält. Die Abkürzungen enden auf „Vd“ statt „V“, ggf. gefolgt von einer Nummer (nicht römisch, sondern arabisch).
- Familie Avsunviroidae
- Gattung Avsunviroid
- Gattung Elaviroid
- Gattung Pelamoviroid
- Familie Pospiviroidae
- Gattung Apscaviroid
- Gattung Cocadviroid mit der Spezies Citrus bark cracking viroid (CBCVd)[18]
- Gattung Coleviroid
- Gattung Hostuviroid
- Gattung Pospiviroid mit der Spezies Potato spindle tuber viroid (PSTVd) und Citrus exocortis viroid (CEVd)
Satelliten
Das ICTV hat im November 2018 erstmals eine Taxonomie auch für Satelliten-Viren (en. Satellite Viruses) herausgegeben, ebenfalls mit demselben Aufbau, jedoch mit Namensbestandteil „satellit“.
- Familie Alphasatellitidae
- Unterfamilie Geminialphasatellitinae
- Gattung Ageyesisatellite
- Gattung Clecrusatellite
- Gattung Colecusatellite (mit den Spezies Tomato leaf curl Buea alphasatellite und Tomato leaf curl Cameroon alphasatellite)
- Gattung Gosmusatellite
- Unterfamilie Nanoalphasatellitinae
- Gattung Clostunsatellite
- Gattung Fabenesatellite
- Gattung Milvetsatellite
- Gattung Mivedwarsatellite
- Gattung Sophoyesatellite
- Gattung Subclovsatellite
- Unterfamilie Petromoalphasatellitinae
- Gattung Babusatellite
- Gattung Cocosatellite
- Gattung Coprasatellite
- Gattung Kobbarisatellite
- Gattung Muscarsatellite
- Unterfamilie Geminialphasatellitinae
- Familie Tolecusatellitidae
- Gattung Betasatellite
- Gattung Deltasatellite
Neben der Familie Sarthroviridae wurde vorgeschlagen, folgende Viren ebenfalls als Satelliten aufzunehmen: Albetovirus, Aumaivirus, Papanivirus und Virtovirus.[19]
Prionen
- Vertebrate Prions (Wirbeltier-Prionen)
- Fungal Prions (Pilz-Prionen)
Die Virus-Taxonomie unterliegt ständigen Veränderungen, die einerseits durch neu entdeckte Viren (speziell in marinen und extremen Lebensräumen) bedingt sind, andererseits dadurch, dass aufgrund immer umfangreicherer Sequenzdaten neue verwandtschaftliche Beziehungen erschlossen werden. In der Regel werden Vorschläge für neue Virustaxa von internationalen Arbeitsgruppen dem ICTV unterbreitet, die allerdings schon vor ihrer offiziellen Übernahme in Publikationen oder Datenbanken, darunter denen des National Center for Biotechnology Information (NCBI) verwendet werden. Früher waren dies in erster Linie serologisch begründete Serogruppen, neuerdings sind es sehr oft MAGs (MAG: Metagenom-assembliertes Genom), mit denen neue Viruarten innerhalb von Gattungen und Familien, aber auch neu geschaffene Virusordnungen, -familien (z. B. „Pithoviridae“) oder -unterfamilien begründet werden. Die Taxonomie des NCBI umfasst neben den offiziellen Virustaxa des ICTV auch solche Vorschläge.[14]
- C. M. Fauquet, M. A. Mayo et al.: Eighth Report of the International Committee on Taxonomy of Viruses. London, San Diego, 2012, ISBN 0-12-249951-4
- A. M. Q. King, M. J. Adams, E. B. Carstens, E. J. Lefkowitz (Hrsg.): Virus Taxonomy. Ninth Report of the International Committee on Taxonomy of Viruses. Amsterdam 2005, ISBN 978-0-12-384684-6
- Eugene V. Koonin, Tatiana G. Senkevich, Valerian V. Dolja: The ancient Virus World and evolution of cells, in: Biol Direct 1:29, 19. September 2006, doi:10.1186/1745-6150-1-29, PMID 16984643, PMC 1594570 (freier Volltext)
- ICTV: Taxonomy Browser, Master Species List
- NCBI: Viruses. Anm.: Das NCBI kennt die taxonomische Rangstufe „Realm“ nicht und führt die Viren in ihrer Gesamtheit (sachlich inkorrekt) als ein Taxon vom Rang Superreich (engl. superkingsom).
- Gattung Deltavirus und ähnliche.
Wikiwand in your browser!
Seamless Wikipedia browsing. On steroids.
Every time you click a link to Wikipedia, Wiktionary or Wikiquote in your browser's search results, it will show the modern Wikiwand interface.
Wikiwand extension is a five stars, simple, with minimum permission required to keep your browsing private, safe and transparent.