Iridoviridae
Familie im Reich Viren (Virus) Aus Wikipedia, der freien Enzyklopädie
Der Begriff Iridoviridae bezeichnet eine Familie von Viren, die der Klasse Megaviricetes in dem Phylum Nucleocytoviricota (veraltet Nucleocytoplasmic large DNA viruses, NCLDV; frühere Vorschläge für dieses Taxon hatten auf „Nucleocytoplasmaviricota“ bzw. – im Rang einer Ordnung – „Megavirales“ gelautet) zugerechnet wird.[1]
| Iridoviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

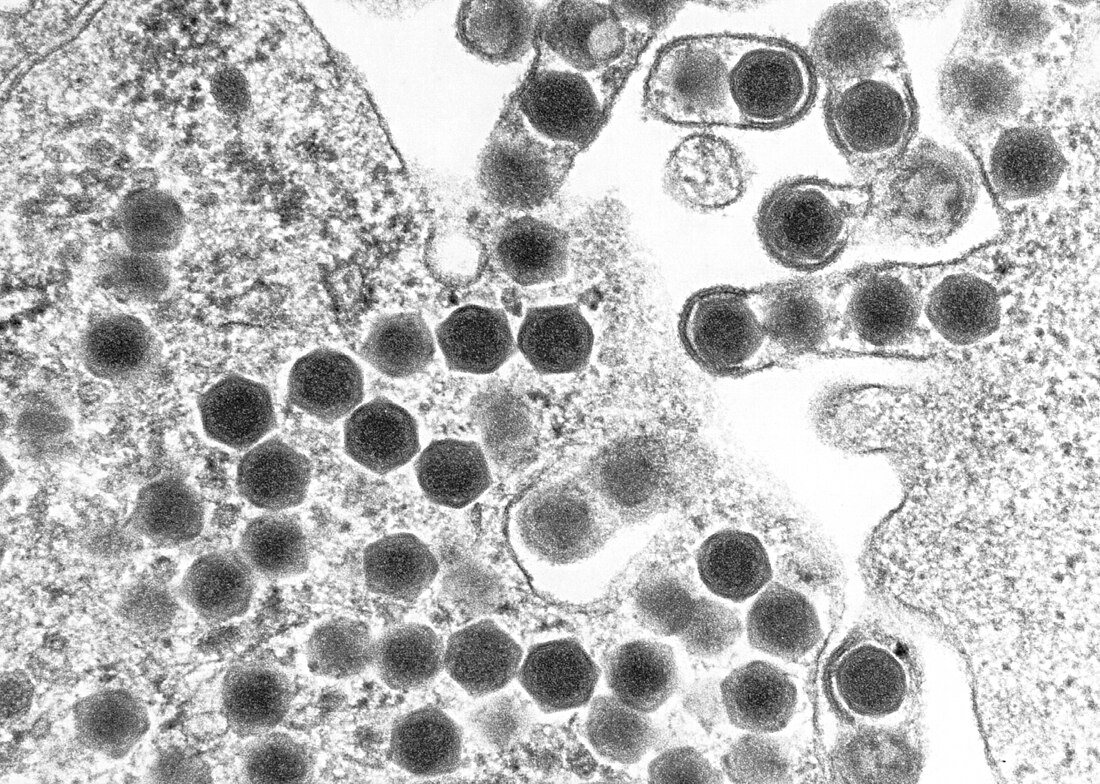
TEM-Aufnahme von Ranaviren (dunkle Sechsecke), | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Iridoviridae | ||||||||||||||
| Links | ||||||||||||||
|
Die Mitglieder der Familie Iridoviridae werden informell als „Iridoviriden“ bezeichnet (en. iridovirids; vgl. Familie Hominidae: Hominiden, en. hominids), im Unterschied zu den Mitgliedern der (Teil-)Gattung Iridovirus, die informall „Iridoviren“ genannt werden (en. iridoviruses).[2]
Systematik
Zusammenfassung
Kontext
Gemäß International Committee on Taxonomy of Viruses (ICTV) ist die innere Systematik wie folgt (Stand MSL#39v1 30. April 2024):[3][4]
Familie Iridoviridae
- Unterfamilie Alphairidovirinae
- Genus Lymphocystivirus (LCDV)[5]
- Genus Megalocytivirus
- Genus Ranavirus
- Unterfamilie Betairidovirinae
- Genus Chloriridovirus
- Genus Daphniairidovirus
- Species Daphniairidovirus daphnia1 (früher Daphniairidovirus tvaerminne)
- Genus Decapodiridovirus
- Species Decapodiridovirus litopenaeus1
- Genus Iridovirus
Weitere Vertreter der Familie wurden vorgeschlagen,[7] darunter
- „Sergestid iridovirus“[6][9]
- „LCIVAC01“ aus Metagenomanalysen vom Schwarzen Raucher Lokis Schloss (englisch Loki’s castle): ein LCV (Loki’s Castle Viruses, iridovirus-like).[10]
- „Acipenser Iridovirus European“ („AcIV-E“)[11][12]
Halaly et al. (2019) schlagen für die Iridoviridae folgendes Kladogramm vor (ergänzt um die Gattungen Daphniairidovirus, und Decapodiridovirus):[13]
| Iridoviridae |
| ||||||||||||||||||||||||||||||
Seit dem Jahr 2000 unterstützen mehrere Studien die Annahme, dass die Ascoviren sich aus den Iridoviridae entwickelt haben.[14][15][16][17] Ein Kladogramm der Asco- und Iridiviridae findet man beim ICTV,[18] und in Fenner’s Veterinary Virology (Fifth Edition) 2017[19] Danach sind die Iridoviridae eine Schwestergruppe der Ascoviridae-Gattung Toursvirus (mit DpTV alias DpAV), während die Gattung Ascovirus eine basale Stellung in der gemeinsamen Klade einnimmt. Nach Andreani et al. (2018) und Chinchar et al. (2020) sind umgekehrt die Ascoviridae eine Schwestergruppe der Alphairidovirinae, während hier die Betairidovirinae basal in der gemeinsamen Klade stehen.[20][21] Diese Ansicht wird auch durch Rolland et al. (2019) unterstützt. Neben den Unterfamilien der Iridoviridae erscheinen die Ascoviridae vom Rang einer Unterfamilie innerhalb einer gemeinsamen Familie „Irido-Ascoviridae“. Die Betairidovirinae sind möglicherweise nicht monophyletisch.[22]
Einige ursprünglich als Vertreter der Iridoviridae vorgeschlagene Kandidaten werden heute anderen Familien zugerechnet, so etwa die Spezies ASF-Virus (Erreger der afrikanischen Schweinepest), jetzt Asfarviridae und die Gruppe der Stör-NCLDVs (SNCLDV), jetzt vorgeschlagene Mitglieder der Mimiviridae und dort offenbar dem Cafeteria-roenbergensis-Virus (CroV) nahestehend.
Virologie
Zusammenfassung
Kontext


Die Viruspartikel (Virionen) haben eine ikosohedrale Symmetrie. Das Virion besteht aus drei Domänen, einem äußeren Kapsid, einer intermediären Lipiddoppelschicht und einem zentralen Core mit DNA-Protein-Komplexen. Gelegentlich kann bei den Viren eine äußere Virushülle identifiziert werden. Dies hängt davon ab, ob die Viren durch Knospung oder nach einer Lyse der Wirtszelle aus dieser heraus gelangen.
Für das Genom der Iridoviridae wird je nach Autor eine Länge von ca. 140–303 bzw. 150–280 kbp (Alphairidovirinae: 100–170 kbp, Betairidovirinae: 135–213 kbp) angegeben.[23] Bei Wiseana iridescent virus beträgt die Genomlänge beispielsweise von 205.791 bp mit vorausgesagt 193 kodierten Proteinen.[24]
Genexpression
Wie bei Herpesviren erfolgt die Transkription in den drei Schritten: „immediate-early“, „delayed-early“ und „late“. Bei jedem Zeitpunkt des Prozesses gibt es Regulationsmechanismen durch Induktion und Produkthemmung.
Replikation
Der Zusammenbau des Virus geschieht im Zytoplasma, ein Teil der Replikation erfolgt im Kern der Wirtszelle. Die Viren dringen in die Zelle ein und verlieren ihre Hülle. Die virale DNA gelangt dann in den Zellkern und wird durch die Pol II transkribiert. Die Synthese von Wirtszellproteinen gelangt zum Stillstand. Die Virus-DNA dient als Vorlage für die DNA-Replikation im Zytoplasma. Dabei werden große Concatamere aus viraler DNA gebildet. Sie werden von Virusproteinen verpackt und durch Knospung oder Lyse aus dem Inneren des Wirtszelle freigelassen.
Pathogenese
Zur Pathogenese der Iridoviren ist wenig bekannt. Sie scheint aber temperaturabhängig zu sein und das Virus daher von poikilothermen Wirten abhängig zu sein.
Wirtsspektrum
Mitglieder der Familie der Iridoviren infizieren hauptsächlich invertebrate Wirte, befallen aber auch Fische, Amphibien und Reptilien.
Siehe auch
- „Namao-Virus“ und die Gruppe „sNCLDV“ (Stör-NCLDV) – vorgeschlagene Mitglieder der Mimiviridae. Einige Kandidaten könnten eher dorthin gehören als zu den Iridoviridae.
Literatur
- V Gregory Chinchar, Thomas B Waltzek, Kuttichantran Subramaniam: Ranaviruses and other members of the family Iridoviridae: Their place in the virosphere. In: Virology, 2017, doi:10.1016/j.virol.2017.06.007
- James K. Jancovich, Natalie K. Steckler, Thomas B. Waltzek: Ranavirus Taxonomy and Phylogeny. In: Ranaviruses. Springer, 2015, ISBN 978-3-319-13755-1, S. 59–70, doi:10.1007/978-3-319-13755-1
Weblinks
Commons: Iridoviridae – Sammlung von Bildern, Videos und Audiodateien
- Iridoviruses. ( vom 10. Januar 2012 im Internet Archive) MicrobiologyBytes
- Viral Bioinformatics Resource Center & Viral Bioinformatics – Canada, University of Victoria (athena.bioc.uvic.ca ( vom 17. August 2007 im Internet Archive))
- The Universal Virus Database of the International Committee on Taxonomy of Viruses ncbi.nlm.nih.gov
- Iridoviridae. Viralzone
Einzelnachweise
Wikiwand - on
Seamless Wikipedia browsing. On steroids.