热门问题
时间线
聊天
视角
核酸双螺旋
由双链分子形成的结构 来自维基百科,自由的百科全书
Remove ads
在分子生物学中,双螺旋[1]是指由核酸(如DNA和RNA)的双链分子所形成的结构。核酸复合物的双螺旋结构是它的二级结构的结果,并且是确定其三级结构的基本组成部分。这个术语因詹姆斯·杜威·沃森于1968年出版的《双螺旋:发现DNA结构的故事》而闻名。

双螺旋结构的聚合物,是由核酸的单体核苷酸的碱基配对在一起[2]在最常见的双螺旋结构物质B-DNA中(见下)中,双螺旋是右手螺旋,环绕一圈大概经过10–10.5 个核苷酸.[3]DNA的双螺旋结构中包含一个大沟和小沟,大沟比小沟宽。[2]因此大多数DNA结合蛋白结合在大沟上。[4]
历史
DNA的双螺旋结构由詹姆斯·杜威·沃森和弗朗西斯·克里克首次发表在1953年的《自然》期刊上。[5] (1954年还发表了三维坐标图[6]) 成果基于罗莎琳·富兰克林1952年拍摄的X射线衍射图 "照片51"[7]和她与雷蒙·葛斯林[8][9]、莫里斯·威尔金斯、艾力克·斯托克斯和贺伯特·威尔森共同拍摄的更为清晰的DNA图像 [10] 以及埃尔文·查戈夫提供的提示碱基配对的化学及生物化学信息。[11][12][13][14][15][16]先前莱纳斯·鲍林曾提出三螺旋模型。[17]
对DNA双螺旋结构的理解有助于阐明碱基配对的机制,以及遗传信息是如何通过碱基配对存储在DNA中并随之复制的,为此,双螺旋结构的发现被视为20世纪最重要的科学发现之一。克里克,,沃森和威尔金斯为此各获1962年诺贝尔生理学或医学奖的三分之一。(富兰克林,她突破性的X-射线衍射数据用于制定的DNA结构,则于1958年去世,因而没有资格被提名。)
Remove ads
核酸的熔解与杂交
核酸杂交是互补碱基配对形成双螺旋结构的过程,而核酸熔解是双链之间作用力被打断,分解为两条单链的过程。这些作用力很弱,容易用温和的加热、酶或物理作用破坏。熔解在核酸的特定位点更易发生[18] :富含T和A的序列比富含C和G的更易熔解. 某些短序列如 T A 和 T G 也更容易熔解。[19] 这个力学现象在生物体本身也有反映,例如基因起始位置的TATA盒就让RNA聚合酶更容易打开DNA双链开启转录。
只要DNA不超过10000碱基对(10kbp),一般都容易用温和的加热分开双链。聚合酶链式反应(PCR)采取的就是这个手段。更长的DNA分子由于双链的缠绕而难以分离。细胞解决这个问题的手段是在解旋酶工作的同时,用拓扑异构酶将DNA的两根磷酸骨架之一的化学键打断,这样一条链就可以绕着另一条链转,从而在解旋酶的作用下与之分离,让阅读DNA序列的酶,例如DNA聚合酶得以前进。
碱基对的空间几何

碱基对的空间构型可以用6个坐标来描述,它们分别是shift, slide, rise, tilt, roll, 和twist。 这些值精确地定义核酸分子中每个碱基或碱基对相对于上一个碱基沿螺旋轴的位置变化和空间朝向。它们共同表征了分子的螺旋结构。 在“正常”结构被破坏的DNA或RNA区域中,这些值的变化可用于描述这种破坏。
核酸链上的相邻碱基的相对几何位置,有以下参数可以考虑:[20][21][22]
- Shear
- Stretch
- Stagger
- Buckle
- Propeller twist: 同一碱基对内的一个碱基相对于另一个碱基的扭转
- Opening
- Shift: 平行于碱基对平面,垂直于碱基氢键方向,沿着小沟向大沟方向的距离。
- Slide: 平行于碱基对平面,沿着一条核酸链向另一条核酸链方向的距离。
- Rise: 沿着双螺旋轴方向的距离
- Tilt: 绕着Shift轴的旋转
- Roll: 绕着Slide轴的旋转
- Twist: 绕着Rise轴的旋转
- vx-displacement
- y-displacement
- inclination
- tip
- 节距(pitch): 双螺旋完整转过一周沿螺旋轴升高的距离
由 Rise 值和 twist 值可以得出核酸的手性和节距(pitch)。 其它的坐标参数可以为零。 Slide 和 shift 值在 B-DNA 中通常较小,在 A- 和 Z-DNA 中较大。非零的 Roll 和 tilt 值意味着相邻的碱基对不平行,它们通常取较小的值。
注意“Tilt”值通常在文献中有不同意义,即碱基对轴相对于螺旋轴垂线的偏移。在基于螺旋的坐标系统中,它被定名为“Inclination”。
螺旋的空间几何


在自然界中发现至少三种DNA构象,即A-DNA,B-DNA和Z-DNA。 詹姆斯·杜威·沃森和弗朗西斯·克里克描述的“B”形式被认为在细胞中占主导地位[23]。它的宽度为23.7Å,每10 bp序列延伸34Å。双螺旋在溶液中每10.4-10.5碱基对围绕其轴旋转一圈。这种扭曲频率(称为螺旋节距,helical pitch)很大程度上取决于每个碱基施加在核酸链上相邻碱基的堆叠力。
A-DNA和Z-DNA的几何形状和尺寸与B-DNA显著不同。 A构象似乎只出现在DNA的脱水样品中,例如在结晶学实验中使用的那些;在DNA和RNA链的杂交配对中,DNA可能也呈A构象。细胞已被甲基化的DNA片段则可采用Z几何结构,以便基因调节。Z构象中,链绕螺旋轴的转向与A-DNA和B-DNA相反。还有证据表明蛋白质-DNA复合物形成Z-DNA结构。
还有其他可能构造。到目前为止,已经描述了A-DNA,B-DNA,C-DNA,E-DNA[24],L-DNA(D-DNA的对映体形式)[25],P-DNA[26],S-DNA,Z-DNA等。事实上,现在只有字母F,Q,U,V和Y可用于描述将来可能出现的任何新的DNA结构[27][28]。然而,大多数这些形式是合成产生的,并且在天然存在的生物系统中没有观察到[來源請求]。还有三链DNA形式和四链体形式,如G-四链体和i-motif。
Remove ads
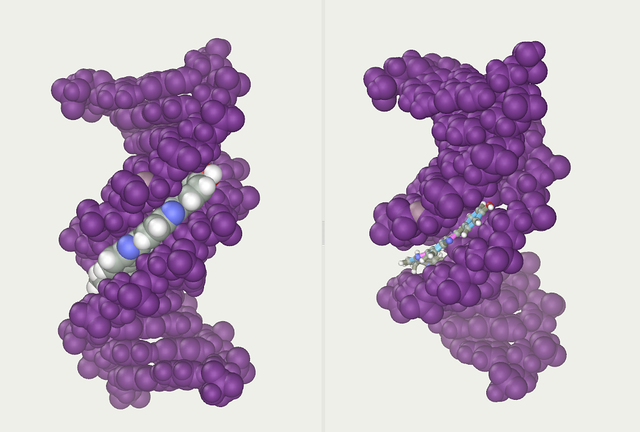
通过描绘双螺旋股线之间的凹槽可以找到另一个双螺旋。这些空隙紧邻碱基对,可以提供结合位点。由于股线彼此不直接相对,因此两条沟的尺寸不一样。 大沟,即主凹槽,宽22Å,而小沟宽度为12Å[32]。小沟的狭窄意味着在大沟种中更容易接近碱基的边缘。结果,可以与双链DNA中的特定序列结合的转录因子等蛋白质通常与暴露在大沟中的碱基侧面接触[4]。DNA处于非典型构象时情况有所差异,但大沟和小沟总是按照DNA被扭转回B构象时的形态被命名。
刚度
DNA是一种相对刚性的聚合物,通常被建模为蠕虫状链。 它有三个重要的自由度:弯曲,扭曲和压缩,每一种都对细胞内DNA的可能性产生特别的限制。 扭曲/扭转刚度对于DNA的环化和DNA结合蛋白相对于彼此的取向是重要的;弯曲/轴向刚度对于DNA包裹和环化以及DNA-蛋白质相互作用是重要的。 在张力不大的情况下,压缩/伸展刚度相对不重要。
弹性
较长的DNA片段在张力下具有熵弹性。当DNA处于溶液中时,由于溶剂的热浴中可用的能量,它经历连续的结构变化。这是由于分子的热振动与水分子的连续碰撞相结合。出于熵的原因,更紧凑的松弛状态比伸展状态更容易,因此DNA分子几乎普遍以纠结的松弛形式存在。出于这个原因,单个DNA分子将在力的作用下伸展,使其变直。使用光镊,从聚合物物理学角度研究和分析了DNA的熵拉伸行为,并且已经发现DNA在生理学上可获得的能量尺度下表现得很像Kratky-Porod蠕虫状链模型。
在足够的张力和正扭矩下,DNA被认为经历了相变,碱基向外张开,磷酸盐向中间移动。这种过度拉伸DNA的结构被称为“P型DNA”,以纪念莱纳斯·鲍林,他最初将其作为DNA的可能结构。[26]
实验证据表明,在没有施加扭矩的情况下拉伸DNA,也会引发一种或一系列相变,导致通常称为S-型DNA的其他结构。尽管已经进行了许多计算机模拟研究(例如[33], [34]),但由于在施加力的情况下难以在溶液中进行原子分辨率成像,这些结构尚未明确地表征。
有些假想的S-DNA结构保留碱基对堆叠和氢键(富含GC),但通过 tilt 释放延伸的结构,还有模型假想碱基堆叠部分熔化,而整体保留碱基-碱基结合的结构(富含AT)。
碱基对堆叠的周期性破裂,每三个bp发生一次断裂(因此每三个bp-bp步骤中有一个)已被提议作为规则结构,其保持基础堆叠的平面性并释放适当的延伸量,[35]术语“Σ-DNA”作为助记符引入,西格玛字符的三个右向点用作三个分组碱基对的提醒。 Σ形式已被证明具有GNC基序的序列偏好,这些基序在GNC_假设下被认为具有进化重要性。[36]
Remove ads
超螺旋和双螺旋拓扑

在没有扭转应变的情况下,DNA形式的B形螺旋扭转360°/ 10.4-10.5bp。但是许多分子生物学过程会引起扭转应变。具有过量或不充分螺旋扭曲的DNA区段分别称为正或负“超螺旋”。体内DNA通常是负超螺旋的,这有利于RNA转录所需的双螺旋的解旋。
在细胞内,大多数DNA在拓扑学上受到限制。 DNA通常存在于拓扑闭合的闭环(例如原核生物中的质粒)中,或者作为非常长的分子,其微小的扩散系数有效地形成了拓扑上闭合的结构域。DNA的线性切片通常也与蛋白质或物理结构(例如膜)结合以形成闭合的拓扑环。
克里克是第一个强调DNA超螺旋环绕数重要性的人。在1976年发表的一篇论文中,克里克概述了如下问题:
在考虑由闭合的双链DNA分子形成的超螺旋时,需要某些数学概念,例如环绕数和扭转数。本文解释了这些概念应用于闭合带的含义,以及闭合曲线的扭力数的含义。本文给出了一些简单的例子,其中一些可能与染色质的结构有关。[37]
对DNA拓扑的分析需要用到以下参数:
- L = 环绕数 - DNA双链中,一条链围绕另一条转过的圈数。对于一个封闭的环,连接数是整数,对于一个拓扑上封闭的区域,它是一个常数。
- T = 扭转数 - 双链DNA螺旋中绕双螺旋轴转过的总转数。假设没有嵌入剂(例如,溴化乙锭)或其他改变DNA刚度的元素,T通常倾向于接近拓扑开放的双链DNA螺旋在溶液中的转数:碱基对数/10.5。
- W = 绞拧数 - 双螺旋绕超螺旋轴转过的总转数。
- L = T + W ,并且 ΔL = ΔT + ΔW
封闭拓扑域中T的任何变化必须通过W的变化来平衡,反之亦然。这导致DNA的更高级结构。具有0绞拧数的圆形DNA分子将是圆形的。如果随后通过超螺旋增加或减少该分子的T,那么将适当地改变W,使得该分子绕结成环形超螺旋。
当一条双链螺旋DNA的两个末端相互连接,形成一个圆时,这些链在拓扑结构上被打结。这意味着两条链不能在任何不涉及链断裂的过程中分离。拓扑异构酶承担了解结的任务。这些酶专用于通过切割一条或两条链来打开环状DNA,使得另一条双链或单链区段可以通过。对于环状DNA和具有类似拓扑约束的线性DNA,其复制和各种类型的重组必需要先解旋。
Remove ads
真核生物细胞基因组中的DNA有多余环绕数,被称为环绕数悖论[38]。它在核小体的结构被判明后得到了解决:在组蛋白八聚体周围缠绕着过度扭转的DNA左手螺旋。[39][40]
参见
- G-四联体
- DNA纳米技术
- 核酸结构
- 《核酸的分子结构:脱氧核糖核酸之构造》(沃森与克里克1953年的论文)
参考文献
Wikiwand - on
Seamless Wikipedia browsing. On steroids.
Remove ads
