热门问题
时间线
聊天
视角
DNA定序
分子生物学技术 来自维基百科,自由的百科全书
Remove ads
DNA定序(DNA sequencing)又稱DNA測序,是指分析特定DNA片段的鹼基序列,也就是腺嘌呤(A)、胸腺嘧啶(T)、胞嘧啶(C)與鳥嘌呤(G)的排列方式。快速的DNA定序方法的出現極大地推動了生物學和醫學的研究和發現。
在基礎生物學研究中,和在眾多的應用領域,如診斷,生物技術,法醫生物學,生物系統學中,DNA序列知識已成為不可缺少的知識。具有現代的DNA定序技術的快速定序速度已經有助於達到定序完整的DNA序列,或多種類型的基因組定序和生命物種,包括人類基因組和其他許多動物,植物和微生物物種的完整DNA序列。
RNA定序則通常將RNA提取後,反轉錄為DNA後使用DNA定序的方法進行定序。目前應用最廣泛的是由弗雷德里克·桑格發明的桑格定序[1]。新的定序方法,例如454生物科學的方法和焦磷酸定序法。

Remove ads
應用
DNA定序可用於確定任何生物的單個基因的序列,較大的遺傳區域(即基因簇或操縱子的簇),完整的染色體或整個基因組。 DNA定序也是對RNA或蛋白質進行定序的最有效方法(通過對開放閱讀框定序)。目前,DNA定序已成為生物學和其他科學領域(如醫學,法醫學或人類學等)的關鍵技術。
在分子生物學中,DNA定序可被用於研究基因組及其編碼的蛋白質。利用定序獲得的資訊,科研人員能夠辨識基因的變化,基因與疾病和表型的關聯,並確定潛在的藥物靶點。
由於DNA是攜帶有遺傳資訊的大分子,在演化生物學中,DNA定序被用於研究不同生物體之間的相關性以及它們是如何演化的。
宏基因組學是一門直接取得環境中所有遺傳物質的研究。環境包括但不限於水體,污水,污垢,從空氣中過濾出的碎片或者從生物體採集的樣本。了解在特定環境中存在哪些生物體對於生態學,流行病學,微生物學和其他領域的研究至關重要。DNA定序使研究人員能夠確定微生物群中可能存在哪些類型的微生物。
醫療人員可通過對患者基因(基因組)的定序結果確定該患者是否有攜帶遺傳性疾病的風險。需要注意的是,該方法屬於基因檢測,有些基因檢測不會用到DNA定序技術。
DNA定序可以與DNA圖譜鑑定(基因指紋分析,英語:DNA profiling)一起用於法醫鑑定和親子鑑定。 DNA測試在過去的幾十年中發展迅猛,目前已能夠做到將DNA鑑定結果與被調查對象聯絡起來。指紋,唾液,毛囊等中的DNA特徵可以將不同的生物體進行區分。測試DNA是一種可以檢測DNA鏈中特定基因組並生成唯一的個性化DNA模型的技術。每一種有機體都有其DNA特徵,並可以通過DNA測試來確定。兩個人具有完全相同的DNA特徵是非常罕見的,因此保證了DNA測試的成功。
歷史

去氧核糖核酸(DNA)最早在1869年由Friedrich Miescher發現並分離出來,但由於當時普遍認為遺傳資訊儲存於蛋白質而不是DNA中,因此在過去幾十年中DNA一直沒有得到充分研究。1944年,由於Oswald Avery,Colin MacLeod和Maclyn McCarty的一些實驗表明,純化的DNA可以將一種細菌變成另一種細菌,這種情況才發生了變化。這也是首次DNA顯示出改變細胞特性的能力。
1953年,James Watson和Francis Crick根據Rosalind Franklin研究的結晶X射線結構提出了他們的雙螺旋DNA模型。根據該模型,DNA由彼此纏繞的兩條核苷酸鏈組成,通過氫鍵連接在一起並以相反方向執行。每條鏈由四個互補的核苷酸組成:腺嘌呤(A),胞嘧啶(C),鳥嘌呤(G)和胸腺嘧啶(T),其中A與T配對,C與G配對。他們提出的這種結構,使得每條單鏈都可被用於重建另一條鏈,並且讓遺傳資訊代代相傳。
對蛋白質進行定序的基礎首先由弗雷德里克·桑格(Frederick Sanger)的工作奠定,他於1955年完成了胰島素(胰腺分泌的一種蛋白質)中所有胺基酸序列的定序工作。這是首個確鑿的證據證明蛋白質是具有特定分子模式的化學實體,而不是懸浮在流體中的隨機混合物。桑格在胰島素定序方面的成功使得X射線晶體學家大為振奮,包括華生和克里克,他們現在正試圖理解DNA如何指導細胞內蛋白質的形成。在1954年10月弗雷德里克·桑格出席一系列講座後不久,克里克開始發展一種理論,認為DNA中核苷酸的排列決定了蛋白質中胺基酸的序列,從而幫助確定蛋白質的功能。他於1958年發表了這一理論。
Remove ads
RNA定序是最早的核苷酸定序形式之一。 RNA定序的主要標誌是1972年和1976年Walter Fiers及其同事在根特大學(根特,比利時)確定並發表的第一個完整基因序列和噬菌體MS2的完整基因組。傳統的RNA定序方法需要建立一個用於定序的互補cDNA(complementary DNA)分子。
確定DNA序列的第一種方法涉及由康奈爾大學的吳瑞於1970年建立的位置特異性引子延伸策略[2]。 DNA聚合酶催化和特定核苷酸標記,這兩者在當前的定序方案中都很重要,用於對λ噬菌體DNA的粘性末端進行定序[3][4][5]。在1970年至1973年間,吳瑞、R Padmanabhan及其同事證明,該方法可用於使用合成的位置特異性引子確定任何DNA序列[6][7][8]。隨後弗雷德里克·桑格(Frederick Sanger)採用這種引子延伸策略在英國劍橋的英國醫學研究委員會(MRC)中心開發了更快速的DNA定序方法,並於1977年發表了「使用鏈終止抑制劑進行DNA定序」的方法。
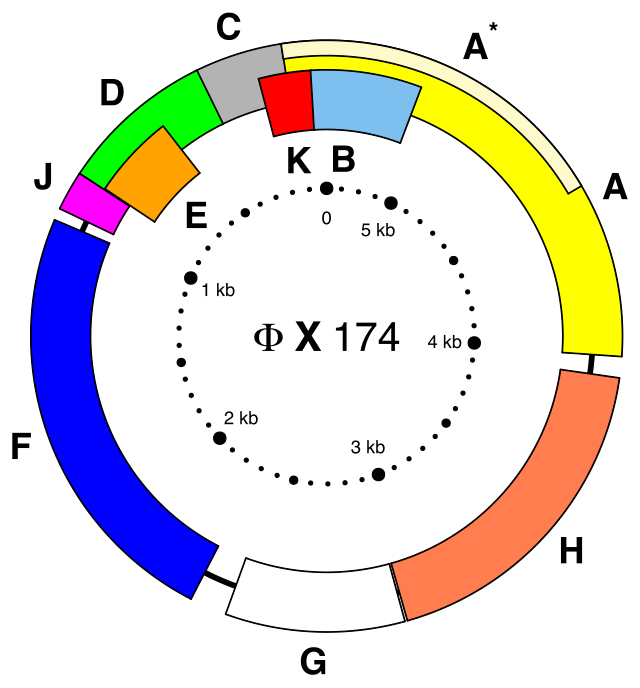
第一個完整的DNA基因組定序是在1977年Φ-X174噬菌體(Phage Φ-X174)的定序工作。醫學研究委員會的科學家在1984年破譯了Epstein-Barr病毒的完整DNA序列,發現它含有172,282個核苷酸。 該序列的完成標誌著DNA定序的一個重要轉折點,它在沒有病毒基因譜知識的情況下實現了DNA定序。
20世紀80年代初,Pohl及其同事開發了一種在電泳時將定序反應混合物的DNA分子轉移到固定基質上的非放射性方法。隨後GATC Biotech公司的DNA定序儀「Direct-Blotting-Electrophoresis-System GATC 1500」商業化,該定序儀在EU基因組定序程式的框架以及酵母釀酒酵母染色體II的完整DNA序列中廣泛使用。加利福尼亞理工學院的Leroy E. Hood實驗室於1986年宣布了第一台半自動DNA定序機。隨後,Applied Biosystems在1987年推出了第一台全自動定序儀ABI 370。以及Dupont公司的Genesis 2000,該儀器使用了一種新的螢光標記技術,可在單一泳道中辨識所有四個雙去氧核苷酸。到1990年,美國國立衛生研究院(NIH)已開始對支原體,大腸桿菌,秀麗隱杆線蟲和釀酒酵母進行大規模定序實驗,費用為每個鹼基0.75美元。同時,人類cDNA序列的定序始於Craig Venter的實驗室,試圖取得人類基因組的編碼部分。 1995年,Venter,Hamilton Smith及其基因組研究所(TIGR)的同事發表了第一個完整的自由生物體細菌流感嗜血桿菌(Haemophilus influenzae)的基因組。該環形染色體中含有1,830,137個鹼基,其在《科學》雜誌中的發表標誌著全基因組鳥槍法定序的首次公開使用,擺脫了初始繪製工作的需要。
Remove ads

定序技術的歷史 [9]
1990年代中後期開發了幾種新的DNA定序方法,並於 2000年在商業DNA定序儀中實施。這些方法統稱為「下一代」或「第二代」定序 (NGS) 方法,以便將它們與包括桑格定序在內的早期方法區分開來。 與第一代定序相比,NGS 技術的典型特徵是高度可延伸,允許一次對整個基因組進行定序。通常,這是通過將基因組片段化成小塊、隨機採樣片段並使用多種技術之一對其進行定序來實現的,例如下面描述的那些。 整個基因組定序是可能的,因為在一個自動化過程中同時對多個片段進行定序(命名為「大規模並行」定序)。
1990年10月26日,錢永健、Pepi Ross、Margaret Fahnestock 和 Allan J Johnston 提交了一項專利,描述了在 DNA 陣列(墨點和單個 DNA 分子)上使用可移除的 3' 阻斷劑進行逐步(「鹼基對鹼基」)定序[10]。 1996 年,斯德哥爾摩皇家理工學院的波爾·尼倫(Pål Nyrén) 和他的學生穆斯塔法·羅納吉(Mostafa Ronaghi)發表了他們的焦磷酸定序方法[11]。
1997年4月1日,Pascal Mayer和Laurent Farinelli 向世界智慧財產權組織提交了描述DNA菌落定序的專利[12]。 本專利中描述的DNA樣品製備和隨機表面聚合酶連鎖反應 (PCR) 陣列方法,與錢永健等人的「鹼基對鹼基」定序方法相結合,現已在Illumina公司的Hi-Seq基因組定序儀中實施。
基本方法
馬克薩姆-吉爾伯特定序(英語:Maxam-Gilbert sequencing)是一項由阿倫·馬克薩姆與沃爾特·吉爾伯特於1976~1977年間開發的DNA定序方法。此項方法基於:對核鹼基特異性地進行局部化學改性,接下來在改性核苷酸毗鄰的位點處DNA骨架發生斷裂[13]。
Sanger(桑格)雙去氧鏈終止法是弗雷德里克·桑格(Frederick Sanger)於1975年發明的。定序過程需要先做一個聚合酶連鎖反應(PCR)。PCR過程中,雙去氧核苷酸可能隨機地被加入到正在合成中的DNA片段里。由於雙去氧核糖核苷酸又少了一個氧原子,一旦它被加入到DNA鏈上,這個DNA鏈就不能繼續增加長度。最終的結果是獲得所有可能獲得的、不同長度的DNA片段。目前最普遍最先進的方法,是將雙去氧核糖核苷酸進行不同螢光標記。將PCR反應獲得的總DNA通過毛細管電泳分離,跑到最末端的DNA就可以在雷射的作用下發出螢光。由於ddATP, ddGTP, ddCTP, ddTTP(4種雙去氧核糖核苷酸)螢光標記不同,電腦可以自動根據顏色判斷該位置上鹼基究竟是A,T,G,C中的哪一個[14]。
進階方法和de novo定序法
霰彈槍定序法(shotgun sequencing,又稱鳥槍法)是一種廣泛使用的為較長DNA定序的方法。它比傳統的定序法快速,但精確度較差。霰彈槍定序法曾經使用於塞雷拉基因組(Celera Genomics)公司所主持的人類基因組計劃。
此章節尚無任何內容,需要擴充。 (2021年2月3日) |
新一代定序
隨著人們對低成本定序的需求與日俱增,推動了高通量定序(high-throughput sequencing)的發展,此技術又稱為二代定序(second generation sequencing)、新一代定序(next-generation sequencing)、次世代定序、大規模平行定序(massively parallel signature sequencing,MPSS),即邊合成邊定序,一次可以對幾十萬至數億條DNA模板同時進行序列測定。這些技術對定序過程采多路復用,同時產生上千或上百萬條序列[15][16]。高通量定序技術的目的是降低DNA定序的成本,這個成本比同樣可實現定序的染料終止法來得低得多[17]。超高通量定序過程中可同時執行高達500,000次的邊合成邊定序[18][19][20]。

454定序法由454生物科學發明,是一個類似焦磷酸定序法的新方法。2003年向GenBank提交了一個腺病毒全序列[33],使得他們的技術成為Sanger定序法後第一個被用來測生物基因組全序列的新方法。454使用類似於焦磷酸定序的方法,有著相當高的讀取速度,大約為5小時可以測兩千萬鹼基對[33]。
正在開發的定序法
高通量定序能一次對幾十到幾百萬DNA分子進行序列測定。
參見
參考文獻
Wikiwand - on
Seamless Wikipedia browsing. On steroids.
Remove ads
