分子生物学
学科 来自维基百科,自由的百科全书
分子生物学(英语:molecular biology)属于生物学的一个分支,是从分子水平上研究生物大分子(蛋白质、核酸等)的结构和功能、生命现象基本过程、分子间相互作用及基因表达调控机制等,以揭示各种生命现象本质的学科。
分子生物学,广义上,是从分子的面向对生物现象的研究;狭义上,是从基因结构和功能的分子层面研究。这是一门从遗传学和生物化学衍生而来的学科。
分子生物学主要致力于开发对细胞中不同系统之间相互作用的研究技术,包括DNA,RNA,蛋白质和蛋白质生物合成之间的关系以及了解它们之间是如何被调控的。
与其他“分子尺度”生物科学的关系
分子生物学的研究者们不仅应用分子生物学特有的技术(参见本条目中“技术”一节),而且越来越多地从遗传学、生物化学和生物物理学的技术和思路中获得启迪,综合利用。因此,这些学科间越来越多地相互融合,不再有明确的分界线。
- “生物化学”主要研究化学物质在生物体关键的生命进程中的作用。生物化学很大程度上专注于生物分子的角色,功能,和结构。生物过程背后的化学性质研究和生物活性分子的合成是生物化学的例子。
- “遗传学”主要研究生物体间遗传差异的影响。这些影响常常可以通过研究正常遗传组分(如基因)的缺失来推断,如研究缺少了一个或多个正常功能性遗传组分的突变体与正常表现型(又称为“野生型”)之间的关系。遗传相互作用(如异位显性)经常会使像基因敲除这类研究的结果难以解释。
- “分子生物学”则主要研究遗传物质的复制、转录和翻译进程中的分子基础。分子生物学的中心法则认为“DNA转录mRNA,mRNA转译蛋白质,蛋白质反过来协助前两项流程,并协助DNA自我复制”;随着研究的深入,发现RNA也可以逆转录成DNA。
在分子生物学中大量工作是定量的,而且最近的许多研究工作是在结合生物信息学和计算生物学的基础之上完成的。从本世纪(二十一世纪)开始,研究基因结构和功能的分子遗传学已经成为发展最快的领域之一。
越来越多的学科已经将目光集中到分子水平的研究中,一方面直接研究相关分子间相互作用,如细胞生物学和发育生物学;另一方面利用分子生物学技术来研究并推测群体和物种的历史贡献(非直接,遗传水平),如进化生物学领域中的群体遗传学和系统发生学。此外,生物物理学除了研究大尺度器官构造之外,一直都有从头研究生物分子的传统。
分子生物学的技术

从1950年代晚期和1960年代早期开始,分子生物学家已经开始学习鉴定、提纯和处理细胞和生物体的分子组分。这些组分包括:DNA,遗传信息的携带者;RNA,作为DNA的“近亲”,既可以是DNA的临时“工作拷贝”,又可以发挥结构分子和酶功能,同时还是蛋白质翻译的结构和功能元件;蛋白质,细胞中的主要的结构分子和酶分子。

分子生物学中最基本的技术是蛋白质的表达和纯化。首先是编码目的蛋白的DNA序列被克隆(用PCR技术和限制性内切酶)到作为表达载体的质粒中。随后构建好的质粒被引入到宿主细胞。编码序列在质粒上的特殊的启动子元件的驱动下,被宿主细胞的表达系统所表达。质粒上通常还带有抗生素抗性标签以便于质粒筛选。[1]
质粒可以被插入到细菌或动物细胞。外源DNA被引入细菌被称为转化(transformation),可以通过电穿孔法、微注射法、正吸收和融合来实现;外源DNA被引入真核细胞,如动物细胞,被称为转染(transfection),转染技术包括磷酸钙法、脂质体法和一些有专利权的商用转染试剂。DNA也可以以病毒或病原菌为载体被带入宿主细胞;应用这种病毒或病菌的转染技术于细胞时,用术语来说就是“对细胞进行转导(transduce)”。
多聚酶链式反应(PCR)是一项用于体外复制DNA的极为通用的技术。简而言之,PCR技术可以使单链DNA被复制数百万次,也允许用事先确定好的方式对被复制的DNA序列进行改动。例如,PCR技术可以用于引入限制性酶切位点,或者对特定的DNA碱基进行突变(改变)。PCR技术还可以用于从cDNA文库获得特定的DNA片段,或者从另一个角度,用于判断一个cDNA文库中是否含有特定的DNA片段。
凝胶电泳是分子生物学最主要的一项技术。其基本原理是:DNA、RNA和蛋白质可以用电场来进行分离。在琼脂糖凝胶电泳中,DNA和RNA可以被琼脂糖凝胶按其分子大小进行分离。同样,蛋白质可以被SDS-聚丙烯酰胺凝胶电泳(SDS-PAGE)按分子量大小分离;此外,蛋白质还可以由于所带电荷的不同被等电聚焦电泳分离。
此方法的命名源自其发明者生物学家Edwin Southern。南方墨点法是探测一个DNA样品中含有特定DNA序列的方法。首先,DNA样品为凝胶电泳所分离;然后,分离的样品通过毛细现象被转移到一张膜上,这一过程被称为“印迹”(blotting)。带有样品的膜就可以用与目标序列互补的标记的DNA标记探针来探测。最初的操作手册大都采用放射性标记,但现在非放射性标记已开始被采用。自从PCR技术被用于检测特定DNA序列后,Southern印迹法在实验室中的应用大为减少。但此方法依然有着其他一些应用,如用于测量转基因鼠的转基因拷贝数,以及用于构建基因敲除的胚胎干细胞系。
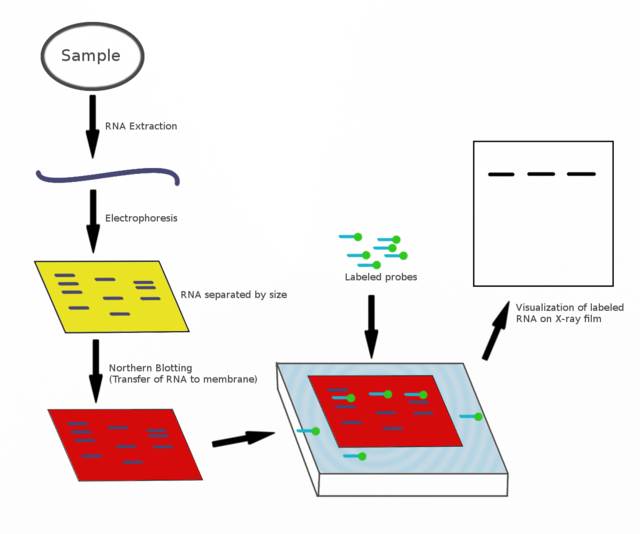
北方墨点法被用于研究特定类别的RNA分子的表达模式(丰度和大小)。与南方墨点法相似,RNA样品为凝胶电泳按大小分离;然后转移到膜上,并用与目标序列互补的标记的探针来探测。实验结果可以根据所用探针的不同以多种方式来观察,但大多数都显示的是样品中被探测的RNA条带的相对位置,也就是分子大小;而条带的强度则与样品中目标RNA的含量相关。这一方法可以测量目标RNA在不同样品中的情况,因此已经被普遍用于研究特定基因在生物体中表达的时刻和表达量,也是这类研究中最基本的手段。
大多数蛋白的抗体可以通过将少量的蛋白注入动物(如鼠、兔、羊)以获得对应注入蛋白的抗体(多克隆抗体)或进一步通过细胞培养获得抗体(单克隆抗体)。这些抗体就可为许多分析和制备技术所使用。在西方墨点法中,蛋白质首先根据分子大小用SDS-PAGE分离。然后将胶中的蛋白质转移到膜(如PDVF膜、尼龙膜或其他可用的膜)上。然后将膜用含有抗体的溶液浸泡,由于抗体可以特异性地结合到目标蛋白上,因此就可以探测膜中的目标蛋白。同样观察结果的方法有很多,包括显色产物、化学发光(chemiluminescence)或放射自显影。一些与西方墨点法相似的方法可以用于直接对细胞和组织中的特定蛋白质进行染色,然而这些被称为“免疫染色”的方法更多的是应用于细胞生物学而非分子生物学。
此外,还有并不常用的“东方墨点法”(对双向电泳后蛋白质分子的印迹分析)、“西南方墨点法”(研究蛋白质和DNA相互作用)和“远端西方墨点法”(Far Western blotting,研究蛋白质-蛋白质相互作用)。
值得一提的是除了南方墨点法的命名是来自于发明者的姓名,其他墨点法的命名则都是源于发明者的幽默:因为南方(Southern)既是墨点法的发明者Edwin Southern的姓,同时其英文含义为“南方”;于是随后发明不同印迹法的研究者们纷纷将这些方法以方位命名,如Northern(“北方”),Western(“西方”)印等等。[2]

DNA阵列是附着于固体支持物的斑点的集合,例如显微镜载玻片,其中每个斑点包含一个或多个单链DNA寡核苷酸片段。阵列使得在一个载玻片上能够放下大量的非常小的(100微米直径)的斑点。每个斑点具有一个DNA片段分子互补的单个DNA序列(类似于南方墨点法)。该技术的变化允许生物在发育的特定阶段的基因表现是合格的(表达谱)。在该技术中,组织中的RNA被分离并转化为标记的互补脱氧核糖核酸(cDNA)。 然后将该cDNA与阵列上的片段杂交,并且可以进行杂交的可视化。由于可以用完全相同的片段位置制备多个阵列,因此它们特别可用于比较两种不同组织(例如健康和癌性组织)的基因表达。此外,人们可以测量什么基因被表达和如何表达随时间或与其他因素的变化而变化。 例如,常见的面包酵母酿酒酵母含有约7000个基因; 使用微阵列,可以定量测量每种基因如何被表达,以及该表达如何变化,例如该表达如何随着温度的变化而变化。有许多不同的方法来制造微阵列; 最常见的是硅芯片,具有~100微米直径的斑点的显微镜载片,定制阵列和在多孔膜(宏阵列)上具有较大斑点的阵列。给定阵列上可以有从100个斑点到超过10,000个斑点。

随着新技术和新方法的不断出现,旧的技术很快就被抛弃。一个很典型的例子就是DNA分子大小的测量:在(琼脂糖/聚丙烯酰胺)凝胶电泳出现之前,DNA分子大小是用蔗糖梯度沉降法来测量的,这一方法费时费力而且花费昂贵;而在梯度沉降法出现之前,黏度法被使用。尽管已经不再为人们所关注,但了解这些过时的技术可能会对解决一些特别的问题有帮助。
历史
在1930年代,由于许多生物化学家发现细胞内的许多分子参与了各种复杂的化学反应,分子生物学由此逐步建立。但直到1938年“分子生物学”一词才由瓦伦·韦弗(Warren Weaver)提出(也有人认为“分子生物学”一词最早于1945年威廉·阿斯特伯里(William Astbury)首先在Harvey Lecture上应用的[3])。瓦伦是当时洛克斐勒基金会自然科学方面的主持人,他相信由于在X射线晶体学等方面的发展,生物学正在进入一个大的转变期,他也因此将基金会的资金用于资助生物领域的研究。
知名的分子生物学家
参见
参考文献
延伸阅读
外部链接
Wikiwand - on
Seamless Wikipedia browsing. On steroids.