Espliceosoma
From Wikipedia, the free encyclopedia
Remove ads
O espliceosoma[1], partícula de splicing ou complexo de empalme é un complexo dinámico de partículas ribonucleoproteicas (compostas de ARN e proteínas) localizado no núcleo celular dos eucariotas.
Remove ads
Características
A súa función é a de asociarse ao pre-ARNm e, por medio de dúas reaccións de transesterificación, procesalo para obter un ARN maduro antes da súa exportación ao citoplasma para ser traducido a proteínas. As diferentes partículas que forman o espliceosoma denomínanse snRNP ou RiboNucleoProteínas nucleares pequenas.
Novas probas derivadas da obtención da primeira estrutura cristalina de intróns do grupo II suxiren que o espliceosoma funciona en realidade como un ribozima, e que usa un mecanismo con dous ións metálicos para a catálise.[2]
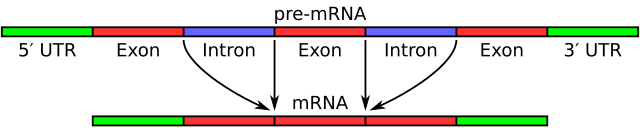
Nos eucariotas é moi frecuente que se encontren nos ARNm en medio das secuencias codificantes ou exóns, outras porcións que non se traducen a proteínas ou intróns. No interior do intrón deben existir un sitio de splicing 3', un sitio de splicing 5' e un punto de ramificación. O sitio de splicing 5' ou sitio doante comprende moi a miúdo unha secuencia GU preto do extremo 5' do intrón. O sitio de splicing 3' ou sitio aceptor termina case sempre cunha secuencia AG. En dirección 5' desde ese AG atópase unha rexión rica en pirimidinas (C e U). Alén desa zona en dirección 5' está situado o punto de ramificación, que contén unha adenina.

A función precisa deste complexo macromolecular que é o espliceosoma é a de recoñecer os sitios de splicing e catalizar a escisión dos intróns dos pre-ARNm e a subseguinte unión dos exóns, proceso chamado splicing.
Remove ads
Tipos de espliceosoma

Existen dous tipos de espliceosomas: o espliceosoma maior, que intervén na maduración da maioría dos ARNm, e o espliceosoma menor, que só intervén na maduración de ARNm cun tipo de intrón pouco frecuente.
- O espliceosoma maior está composto por cinco snRNP : a snRNP U1, a snRNP U2, a di-snRNP U4/U6 e a snRNP U5. Estas partículas están formadas por un pequeno ARN (snRNA) ao que están asociadas proteínas. Distínguense dúas familias destas proteínas:
- 1. As sete proteínas Sm (SmB/B', D1, D2, E, F, G) chamadas proteínas centrais ou core, que se asocian formando un anel pentamérico arredor dos snRNA U1, U2, U4 e U5.
- 2. As proteínas chamadas "específicas" que se asocian unicamente cun tipo determinado de snRNP (como as proteínas U1A, 70K e U1C, específicas da snRNP U1).
- Actualmente coñécense unhas 150 proteínas diferentes que se asocian coas snRNP para formar o espliceosoma.
- O espliceosoma menor funciona de modo similar pero os intróns sobre os que actúa son máis raros e diferentes dos intróns espliceosomais típicos antes descritos, porque teñen sitios de splicing diferentes. As secuencias recoñecidas neste caso son, respectivamente, AC en 5' e UA en 3'. Ademais, só a snRNP U5 é a mesma nos dous espliceosomas; as outras denomínanse análogas funcionais, e son: U11 (con función análoga a U1), U12 (análoga de U2), U4atac (de U4) e U6atac (de U6).
Remove ads
Ensamblaxe do espliceosoma

O modelo de formación dun espliceosoma activo implica unha ensamblaxe ordenada e por fases das snRNP individuais sobre o pre-ARNm. O primeiro recoñecemento do pre-ARNm faino a snRNP U1, que se une ao sitio de splicing do extremo 5' do pre-ARNm xunto con outros factores non asociados a snRNP, para formar o complexo de compromiso (commitment) ou complexo temperán E nos mamíferos.[3][4] Este complexo é un complexo independente do ATP que encamiña ao pre-ARNm ao splicing.[5] A snRNP U2 é agora recrutada na rexión de ramificación por medio de interaccións co compoñente U2AF do complexo E (ou factor auxiliar da snRNP U2) e posiblemente coa snRNP U1. Por medio dunha reacción dependente do ATP, a snRNP U2 asóciase moi estreitamente coa secuencia do punto de ramificación para formar o complexo A. Entón un dúplex formado entre a snRNP U2 e a rexión de ramificación do pre-ARNm fai sobresaír a adenosina do punto de ramificación, marcándoa como o nucleófilo para a primeira transesterificación que vai ter lugar.[6]
A presenza dun residuo de pseudouridina na snRNA U2, case en fronte do punto de ramificación, orixina unha conformación alterada do dúplex ARN-ARN durante a unión da snRNP U2. Especificamente, a estrutura alterada do dúplex inducida pola pseudouridina sitúa o grupo 2' OH da adenosina que sobresae nunha posición favorable para o primeiro paso do splicing.[7] Recrútase agora o grupo de tres snRNP U4/U5/U6 para ensamblarse no espliceosoma en formación, orixinando o complexo B, e despois de sufrir varias reordenacións, o complexo C (é dicir, o espliceosoma) actívase para a catálise.[8][9] Non está claro como se recrutra o trío de snRNP no complexo A, pero este proceso pode estar mediado por interaccións proteína-proteína e/ou interaccións entre bases dos snRNA U2 e U6.
A snRNP U5 interacciona con secuencias dos sitios de splicing 5' e 3' por medio do bucle invariante do snRNA U5[10] e os compoñentes proteicos U5 interaccionan co sitio de splicing 3'.[11]
Durante o recrutamento do trío de snRNP, teñen lugar varias reordenacións ARN-ARN, que preceden ao primeiro paso catalítico, e ocorren máis reordenacións no espliceosoma activo cataliticamente. Varias das interaccións ARN-ARN son mutuamente excluíntes; porén, non se sabe que desencadea estas interaccións, nin a orde destas reordenacións. A primeira reordenación é probablemente o desprazamento da snRNP U1 do sitio de splicing 5' e a formación da interacción de snRNA U6. Sábese que a snRNP U1 está só feblemente asociada cos espliceosomas completos,[12] e a snRNP U1 é inhibitoria para a formación da interacción de U6 co sitio de splicing 5' como se viu en experimentos cun modelo de substrato oligonucleotídico que contiña un curto exón 5' e a secuencia do sitio de splicing 5'.[13] A unión da snRNP U2 á secuencia do punto de ramificación é un exemplo de interacción ARN-ARN que despraza unha interacción proteína-ARN. No momento do recrutamento da snRNP U2, a proteína de unión á ramificación SF1 no complexo E é desprazada, xa que o sitio de unión do snRNA U2 e SF1 son sucesos mutuamente excluíntes.
No snRNA U2 hai outras reordenacións mutuamente excluíntes que se producen entre conformacións alternativas. Por exemplo, na forma activa, o bucle IIa está favorecido; na forma inactiva predomina unha interacción mutuamente excluínte entre o bucle e a secuencia que está en dirección 3'.[9] Non está claro como U4 é desprazada do snRNA U6, aínda que o ARN estivo implicado na ensamblaxe do espliceosoma, e podería funcionar desenrolando U4/U6 e promovendo a formación dunha interacción entre as snRNA U2/U6. As interaccións dos bucles I e II de U4/U6 disócianse, e a rexión do bucle II libre de U6 dóbrase sobre si mesma para formar un bucle intramolecular e xa non se require a U4 para ulteriores ensamblaxes do espliceosoma. A rexión do bucle I libre de U6 únese por emparellamento de bases co snRNA U2 formando a hélice I de U2/U6. Non obstante, a estrutura en hélice I é mutuamente excluínte coa metade 3' dunha rexión en bucle 5' interna do snRNA U2.
Remove ads
Funcionamento do espliceosoma
Os compoñentes do espliceosoma están separados no núcleo e non se ensamblan ata que o espliceosoma empeza a actuar. Esta ensamblaxe faise sobre as secuencias intrónicas de recoñecemento (os sitios de splicing) do pre-ARNm e ten lugar de maneira secuencial, formándose sucesivamente os seguintes complexos:

- Complexo E: O snRNA U1 asóciase ao sitio Guanosil-Uridil do extremo 5' (5'GU) do intrón.
- Complexo A: O snRNA U2 fíxase sobre o punto de ramificación situado a unha distancia de 20-40 nucleótidos do par AC que forma o extremo 3' do intrón.
- Complexo B1: Os snRNA U4 e U6 asócianse entre si e despois o snRNA U5 únese a eles, formando o complexo U4/U5/U6. U6 fíxase sobre U2 mentres que U5 se fixa ao extremo 3' do exón próximo a U1 (o espliceosoma está agora completo), aproximando os extremos do intrón que deben ser escindidos.
- Complexo B2: Libérase U1 do espliceosoma, U5 esvara sobre o intrón e U6 fíxase ao extremo 5' do sitio de escisión.
- Complexo C1: Libérase U4, e pola súa parte U6 e U2 catalizan a reacción de transesterificación e córtase o extremo 5' do intrón. Fórmase unha estrutura con forma de lazo ou bucle chamada lariat, xa que o extremo cortado se une ao punto de ramificación.
- Complexo C2: o extremo 3' do intrón córtase agora, o que libera o intrón, xa que foi cortado polos dous extremos. Ten forma de lazo e será degradado. Despois líganse os dous exóns. Finalmente, o complexo disóciase.
Remove ads
Notas
Véxase tamén
Wikiwand - on
Seamless Wikipedia browsing. On steroids.
Remove ads