Loading AI tools
De Wikipédia, l'encyclopédie libre
Une puce à ADN est un ensemble de molécules d'ADN fixées en rangées ordonnées sur une petite surface qui peut être du verre, du silicium ou du plastique. Cette biotechnologie récente permet d'analyser le niveau d'expression des gènes (transcrits) dans une cellule, un tissu, un organe, un organisme ou encore un mélange complexe, à un moment donné et dans un état donné par rapport à un échantillon de référence.
Les puces à ADN sont aussi appelées puces à gènes, biopuces ou par les termes anglais « DNA chip, DNA-microarray, biochip ». Les termes français microréseau d'ADN et micromatrice d'ADN sont aussi des termes proposés par l'Office québécois de la langue française[1].
Le principe de la puce à ADN repose sur la propriété que possède l'ADN dénaturé (simple brin) de reformer spontanément sa double hélice lorsqu'il est en présence d'un brin complémentaire (réaction d'hybridation). Les quatre bases nucléiques de l'ADN (A, G, C, T) ont en effet la particularité de s'apparier deux à deux par des liaisons hydrogène (A = T et T = A ; G ≡ C et C ≡ G).
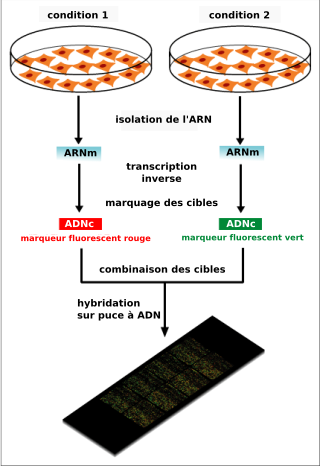
On parle de sonde (fragment d'ADN synthétique représentatif des gènes dont on cherche à étudier l'expression, fixé de façon covalente à la surface de la biopuce) et de cible (ARNm que l’on cherche à identifier et/ou à quantifier (échantillon)). Les cibles sont marquées par fluorescence (voir plus bas).
Les puces à ADN peuvent être utilisées pour mesurer/détecter les ARN qui seront traduits ou non en protéines. Les scientifiques parlent d'analyse d'expression ou de profil d'expression. Puisqu'il est possible de fixer jusqu'à un million de sondes sur une biopuce, les puces à ADN constituent ainsi une approche massive. Elles ont contribué à la révolution de la génomique[2], puisqu'elles permettent, en une seule expérience, d'avoir une estimation sur l'expression de plusieurs dizaines de milliers de gènes. Il existe également un grand nombre d'applications différentes qui font intervenir la technologie des puces à ADN (criblage de mutations, reséquençage, interactions ADN/protéine, écologie microbienne).
En pratique, les ARN totaux sont extraits des cellules étudiées et subissent parfois une amplification qui va permettre d'obtenir une quantité de matériel génétique suffisante pour l'expérience. Ces ARNm sont ensuite transformés en ADN complémentaires (ADNc) par la technique de rétrotranscription et marqués. Ce marquage est aujourd'hui assuré par une molécule fluorescente (fluorochrome). Il existe deux fluorochromes majoritairement utilisés : la Cyanine 3 (Cy3) qui fluoresce dans le vert et la Cyanine 5 (Cy5) qui fluoresce dans le rouge. Les cibles ainsi marquées (ADNc) sont ensuite mises en contact avec la puce (portant l'ensemble des sondes à sa surface), cette étape est nommée hybridation. Chaque brin d'ADNc va alors s'hybrider aux sondes qui lui sont complémentaires pour former un duplex sonde/cible double brin. La biopuce est ensuite lavée par des bains spécifiques pour éliminer les brins d'ADNc ne s'étant pas hybridés car non complémentaires des sondes fixées sur la lame. La biopuce va ensuite être analysée par un scanner à très haute résolution, et ce à la longueur d'onde d'excitation de la Cyanine 3 ou de la Cyanine 5. L'image scannée est alors analysée informatiquement afin d'associer une valeur d'intensité à chaque sonde fixée sur la biopuce et ainsi de déterminer s'il y a eu une hybridation pour chaque sonde.

Lors d'une expérience de biopuce ADN visant à comparer l'expression des gènes entre deux conditions (par exemple : cellule saine versus cellule malade), les ARN totaux des deux populations de cellules sont extraits et marqués chacun avec un fluorochrome différent. Les deux échantillons sont alors déposés simultanément sur la biopuce, on parle d'hybridation compétitive. Pour un gène donné, si le nombre de molécules d'ADNc correspondant à ce gène est plus important dans une condition que dans l'autre, l'hybridation entre ces ADNc et les sondes associées sera favorisée. Ainsi, après avoir scanné la biopuce aux deux longueurs des deux fluorochromes utilisés, il est possible de comparer l'intensité du signal vert et celle du signal rouge et donc de savoir, pour chaque gène étudié, dans quelle condition il est le plus exprimé.
Les balbutiements des puces à ADN se font dès les années 1991 avec une publication[3] de Stephen Fodor (scientifique américain et fondateur de la société Affymetrix) sur la technologie de synthèse in situ. S'ensuivent ensuite plusieurs événements qui entrent en ligne de compte :
On distingue deux types d'analyses de biopuces :
Une des forces de la biopuce à un canal réside dans le fait qu'un échantillon aberrant n'affecte pas l'analyse des autres échantillons. A contrario, chez les biopuces à deux canaux, il suffit d'un seul échantillon de mauvaise qualité pour diminuer drastiquement la qualité des résultats, même si l'autre échantillon cible est parfait. Une autre force est que les données d'une biopuce à un canal sont plus facilement comparées à d'autres biopuces provenant de différentes expériences. De plus, la biopuce à un canal est parfois la seule solution possible pour certaines applications.
Le tableau suivant est repris de la page anglophone de Wikipédia.
| Nom du procédé ou de la technologie | Description |
|---|---|
| Expression des gènes | Il s'agit de l'utilisation la plus connue de la puce à ADN. On compare l'expression des gènes entre différentes conditions données ou au cours du temps. Grâce à l'hybridation et à une analyse d'image effectuée ensuite, ce procédé permet d'identifier quels gènes sont sur- ou sous-exprimés dans une condition donnée. Une fois ces gènes identifiés, d'autres analyses in silico sont nécessaires, telles que des analyses de clustering pour regrouper les gènes présentant le même profil d'expression. Enfin, les résultats seront souvent confirmés gène par gène par des méthodes telles que la PCR quantitative ou le Northern Blot. (méthodes d'analyse des gènes) |
| Immunoprécipitation de chromatine | Les biopuces peuvent aussi utiliser le phénomène d'immunoprécipitation de chromatine (Chromatin immunoprecipitation on Chip, ou ChIP-On-Chip). Ce procédé permet de déterminer la localisation du site de liaison de la protéine dans le génome. |
| Polymorphisme | La puce à ADN peut aussi permettre de détecter du polymorphisme, c'est-à-dire d'identifier des polymorphismes ponctuels d’allèles au sein d’une population ou entre populations, prédire le développement de maladies au sein d'une population, évaluer les mutations ou encore analyser les liens entre gènes. |
| Tiling | Ces puces sont destinées à la recherche de nouveaux transcrits (on entend par là gène transcrit). C'est-à-dire que chaque segment de chromosome (pas seulement les gènes connus) est ciblé par une sonde. Un des intérêts est de découvrir de très nombreux ARN non codants (comme les ARN longs non-codants par exemple). On peut ainsi réussir à cartographier les transcrits, rechercher des exons, ou encore rechercher les facteurs de transcription. |
| Biopuce à gène chimère | Il existe aussi des biopuces à gène chimère (ou gène de fusion). Le principe derrière est celui de l'alternative splicing en anglais, ou encore épissage alternatif en français. Une telle biopuce peut alors détecter les gènes transcrits par fusion, donc de spécimens de cancer. |
| Hybridation génomique comparative | Une puce d'hybridation génomique comparative est une puce à ADN employée dans le but d'analyser les variations du nombre de copies dans l'ADN. Cette technique est principalement utilisée pour diagnostiquer les cancers et les maladies génétiques. |
| GeneID | Ces puces à ADN sont utilisées pour détecter certains types d'organismes dans la nourriture (comme des OGM), des mycoplasmes en culture cellulaire, ou certains agents infectieux pour diagnostiquer des maladies. La technique repose principalement sur la réaction en chaîne par polymérase. |
La liste ci-dessous n'a pas vocation à être exhaustive, mais elle donne un panorama des domaines d'applications des biopuces.
La puce est une lame, généralement en verre, de petite taille environ (6 cm x 3 cm), sur laquelle sont fixés des sondes complémentaire d'un fragment d'acide nucléique (ADN ou ARN) ciblé. Jusqu'à un million de sondes peuvent être fixés sur une puce permettant ainsi l'analyse de plusieurs dizaines voire centaines de milliers de gènes.
Quels types de sonde poser sur la puce[10] ?
| Oligonucléotides | Sondes longues | |
|---|---|---|
| Taille | 15 - 70 mers[alpha 1] | > 100 mers[alpha 1] |
| Avantages | - Détection des SNP (polymorphisme)
- Livré « prêt à spotter » |
- Insensibles aux allèles/variants
- Production en grande quantité - Collections disponibles (EST, BAC...) - Double brin, plus de choix de marquage, meilleur couverture du génome |
| Inconvénients | - Sensibles aux allèles/variants
- Coût de la production de masse - Le design des oligos est une étape complexe |
- Une résolution plus faible
- La production des produits de PCR est lourde - Problème d’hybridation croisée - Erreurs des collections (EST) |
Quels types de gènes sont concernés ?
En soi, aucune contre-indication n'existe concernant le type de gènes à tester (gènes aux fonctions connues ou inconnues). Néanmoins, afin de pouvoir tirer des informations fiables des expériences de biopuces, il est conseillé d'inclure des sondes de contrôle positif et négatif afin de vérifier et contrôler le bon déroulement de chaque étape de l'expérience.
Ce procédé utilise des sondes longues (une centaine de nucléotides) déposées sur la lame. Les sondes sont synthétisées avant d'être déposées sur la surface par rangées d’infimes gouttelettes, ou spots, (d'où le terme anglais microarray, « microtableau »). On utilise des aiguilles fines contrôlées par un bras robotisé qui est plongé dans les spots. Les aiguilles vont alors injecter dans chaque spot les sondes en excès. La «grille» finale représente les profils d’acides nucléiques des sondes préparées et chaque sonde est prête à s'hybrider avec les cibles.
Cette méthode est la plus simple, ce qui rend cette solution plus accessible aux laboratoires académiques. Elle est utilisée par les scientifiques et chercheurs dans le monde entier pour produire les puces adaptées à leur besoins. Les puces sont facilement personnalisées pour chaque expérience car les chercheurs peuvent choisir le type et l'emplacement des sondes, voire synthétiser les sondes eux-mêmes.
Les scientifiques peuvent ensuite générer leurs propres échantillons cibles, utilisés pour l'hybridation avec les sondes, pour enfin analyser les puces avec leurs propres équipements. Cela fournit une puce moins chère (en évitant les coûts d'achat de puces commerciales) et aussi adaptée à leurs exigences.
Les puces qui sont fabriquées de cette manière ne peuvent cependant pas posséder la même densité de sondes que les puces fabriquées par synthèse in situ.

Ces puces sont fabriquées en synthétisant les sondes d'ADN de petite taille (< 80-mers[alpha 1]) directement sur le support (biopuce) par synthèse chimique.
Une image haute résolution est obtenue grâce à des scanners très haute résolution (de l'ordre du micromètre) qui permettent de révéler les interactions sonde/cible par excitation des fluorochromes portés par les cibles. En France la société INNOPSYS développe, fabrique et commercialise une gamme complète de scanners de fluorescence destinés à la lecture et l'analyse de ce type de lames : InnoScan 710 (2 couleurs, 3 µm/pixel, InnoScan 710-IR, InnoScan 910 (2 couleurs, 1 µm/pixel) et InnoScan 1100 (3 couleurs, 0.5 µm/pixel).
Des logiciels interprètent l'intensité des pixels de l'image afin d'en déduire une mesure numérique de l'expression de chaque gène.

De grands volumes de données sont générés par l'analyse d'une puce à ADN, et de nombreuses techniques sont utilisées pour interpréter les résultats de l'expérience. Ces techniques incluent :
Seamless Wikipedia browsing. On steroids.
Every time you click a link to Wikipedia, Wiktionary or Wikiquote in your browser's search results, it will show the modern Wikiwand interface.
Wikiwand extension is a five stars, simple, with minimum permission required to keep your browsing private, safe and transparent.