Filoviridae
famille de virus De Wikipédia, l'encyclopédie libre
La famille des Filoviridae, ou filovirus, appartient à l'ordre des Mononegavirales (mononégavirus) regroupant les virus à ARN monocaténaire non segmenté à polarité négative. Les filovirus sont ainsi dénommés car ils présentent une apparence filamenteuse.
La plupart d'entre eux (mais il existe peu d'exceptions[4]) sont des agents infectieux responsables de fièvres hémorragiques[5] aiguës particulièrement létales, notamment :
- le virus Ebola, responsable de la maladie à virus Ebola ;
- le virus de Marburg, responsable de la fièvre hémorragique de Marbourg.
Taxinomie
Résumé
Contexte
Ils appartiennent à l'ordre des Mononegavirales, comprenant les virus à ARN monocaténaire non segmenté à polarité négative. Initialement classés parmi les rhabdovirus, les filovirus forment aujourd'hui une famille distincte et seraient en réalité plus proches des paramyxovirus[6], parmi lesquels on trouve notamment les virus des oreillons et de la rougeole.
On connaît cinq virus distincts[7],[8], que l'ICTV rattache chacun à l'une des cinq espèces du genre Ebolavirus. Cependant, la taxinomie des filovirus est récente et continue d'évoluer au gré des avancées phylogénétiques, d'où une relative confusion entre les différentes dénominations retenues selon les auteurs[9]. Un usage bien ancré dans les laboratoires fait du virus Ebola une désignation synonyme du genre Ebolavirus décliné en cinq sous-types de virus[10],[11], tandis que la nomenclature adoptée par l'ICTV, faisant du virus Ebola le virus de l'espèce type du genre Ebolavirus, n'a pas encore été ratifiée.

On distingue :
- le virus Ebola proprement dit (EBOV), de l'espèce ebolavirus Zaïre (autrefois ZEBOV), ou sous-type Ebola Zaïre, identifié pour la première fois en 1976 au Zaïre[12] (aujourd'hui République démocratique du Congo) — c'est le plus virulent des cinq virus, à l'origine de l'épidémie de 2014 en Afrique de l'Ouest[13] ;
- le virus Soudan (SUDV), de l'espèce ebolavirus Soudan (autrefois SEBOV), ou sous-type Ebola Soudan, endémique au Soudan du Sud et en Ouganda[14] ;
- le virus Reston (RESTV), de l'espèce ebolavirus Reston (autrefois REBOV), ou sous-type Ebola Reston, identifié en 1983 dans la région de Reston, aux États-Unis[15] ;
- le virus Forêt de Taï (TAFV), de l'espèce ebolavirus Forêt de Taï, autrefois ebolavirus Côte d'Ivoire (CIEBOV), ou sous-type Ebola Forêt de Taï (ou encore Ebola Côte d'Ivoire), identifié en 1994 dans le parc national de Taï, en Côte d'Ivoire, aux confins de la Guinée et du Liberia[16] ;
- le virus Bundibugyo (BDBV), de l'espèce ebolavirus Bundibugyo (autrefois BEBOV), ou sous-type Ebola Bundibugyo, identifié en 2008 dans la région de Bundibugyo, en Ouganda[17].
Virions
Les filovirus sont, comme leur nom l'indique, des particules virales d'apparence filamenteuse. La capside en elle-même présente une apparence filamenteuse en U, en 6, en crochet ou en bâtonnet, et peut être ramifiée[9].
Par ailleurs ce sont des virus enveloppés — la capside n'est pas nue mais recouverte d'une membrane lipidique. Les virions sont également caractérisés par la présence d'une glycoprotéine fortement glycosylée constituée de deux sous-unités différentes et formant des trimères insérés dans l'enveloppe virale en formant des projections de 7 nm espacées d'environ 10 nm[9].
Libération de particules de virus de Marburg
(tomographie électronique)
(tomographie électronique)
Particules filamenteuses (24 h après infection)
Particules sphériques(4 j après infection)
La coloration met en évidence des membranes et le cytoplasme (jaune) et les particules virales (bleu), parfois entourées de vésicules dérivées des cellules et de débris.
Génomique
Résumé
Contexte
Le nom des espèces virales validé par l'ICTV a sensiblement évolué depuis l'identification de ces virus[19],[20],[21],[22],[23],[24]. La taxonomie de la famille peut encore évoluer au fur et à mesure des études génétiques et de l'éventuelle découverte de nouveaux membres de la famille [25], en zone tropicale, mais aussi en zone tempérée (un virus proche d'Ebola a par exemple été trouvé en Europe[26]).
Ces virus possèdent un génome de 19 kilobases, plus long que la plupart des mononégavirus (dont le génome mesure entre 8 et 16 kb). Leur ARN génomique contient un ou plusieurs chevauchements de gènes. Les gènes de ce génome sont organisés selon le schéma 3'-UTR–NP–VP35–VP40–GP–VP30–VP24–L–5'-UTR ; la protéine VP24 est spécifique aux filovirus tandis que la protéine VP30 est partiellement analogue à une protéine uniquement exprimée chez les pneumovirus[9].
Les variations génétiques entre les différents virus du genre Ebolavirus peuvent être illustrées par la comparaison de la taille des différents génomes et leurs taux de GC respectifs (voir tableau ci-contre à droite).[réf. nécessaire]
Par comparaison, parmi les autres filovirus, le virus de Marburg, du genre Marburgvirus, a un génome long de 19,11 kb avec un taux de GC de 38,8 %[27] tandis que le virus Lloviu, du genre Cuevavirus, a un génome long de 18,93 kb avec un taux de GC de 46,0 %[28].
Pouvoir pathogène
Résumé
Contexte
Ces virus se développent dans l’organisme de certains animaux (en particulier chiroptères et primates -- non-humain comme humains [29]).
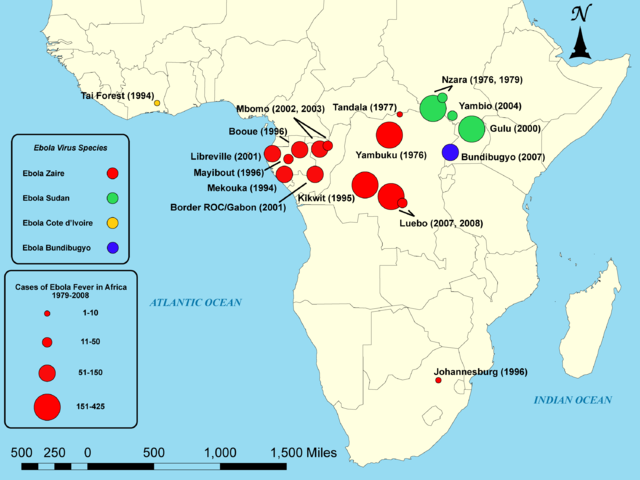
- virus Ebola en rouge ;
- virus Soudan en vert ;
- virus Forêt de Taï en orange ;
- virus Bundibugyo en bleu.
La nature pathogène des différents filovirus, qu'il s'agisse du genre Ebolavirus ou du genre Marburgvirus, est très semblable dans la mesure où ces virus ont tous été associés à des flambées de fièvres hémorragiques chez l'homme et les autres primates avec des symptômes identiques. Ils diffèrent en revanche du point de vue génétique, avec une séquence nucléotidique pouvant varier de 30 à 40 % d'une souche à l'autre, ce qui se traduit par une sévérité très différente entre les pathologies induites chez l'homme par ces différents virus — la létalité peut ainsi être nulle chez les humains pour le virus Reston, mais approcher 90 % pour le virus Ebola — bien que des facteurs environnementaux puissent également expliquer ces différences.[réf. nécessaire]
Le virus Reston a été isolé en 1989 chez des macaques crabiers aux Philippines. Présent également en Chine, il est moins pathogène chez les primates non humains et l'on pensait qu'il n'affectait pas les humains jusqu'à ce qu'on identifie une transmission du porc à l'homme en 2009[31].Le virus Bundibugyo, découvert en 2008, s’apparente davantage au virus Forêt de Taï[7], mais est plus virulent que ce dernier.
Évolution
Résumé
Contexte
Pour savoir si les filovirus sont réellement source d'un risque pandémique [32], il est utile de connaitre l'histoire de ces virus et de déterminer et comprendre leurs capacités évolutives et adaptatives (par le jeu des mutations) à long et moyen terme, et à court terme lors d'une épidémie, ou d'évaluer leur capacité d'adaptation à d'autres environnement et à d'autres hôtes[33].
Selon les données génétiques disponibles, les virus des groupes Marburg (marburgvirus) et Ebola (ebolavirus) du Soudan partageraient un ancêtre commun récent (aux échelles de l'évolution); ils seraient nés de cet ancêtre approximativement 700 ans avant nos jours pour le premier groupe et il y a 850 ans environ pour le second. Et les membres connus de la famille des Filoviridae (dont le virus Lloviu récemment décrit) partageraient un ancêtre commun vieux d'environ 10 000 ans (fin de la dernière glaciation)[34].
Selon Serena A. Carroll & al., comprendre le passé et le paléoenvironnement de ces virus pourraient aider à mieux comprendre le rôle des espèces réservoir et à mieux combattre ou prévenir les épidémies humaines en comprenant mieux les mécanismes d'émergence, l'évolution de la pathogénicité des virus et des risques pour la santé publique et la santé environnementale (écoépidémiologie)[34].
En particulier, il est important de comprendre si et comment des animaux sauvages (viande de brousse, singes destinés à l'expérimentation animale...) ou certains animaux d'élevage ou domestiqués susceptibles de faire l'objet d'échanges internationaux peuvent être porteurs, voire porteurs sains de virus de cette famille, et comment ils lui résistent si c'est le cas, y compris pour le porc par exemple[35] qui est de plus en plus génétiquement homogénéisé dans le monde, un vecteur potentiel de nombreux virus.
Liste des genres et des espèces
Selon l’ICTV (version d'août 2023[3]) :
- genre Cuevavirus
- espèce Cuevavirus lloviuense
- genre Dianlovirus
- espèce Dianlovirus menglaense
- genre Loebevirus
- espèce Loebevirus percae
- genre Oblavirus
- espèce Oblavirus percae
- genre Orthoebolavirus
- espèce Orthoebolavirus bombaliense
- espèce Orthoebolavirus bundibugyoense
- espèce Orthoebolavirus restonense
- espèce Orthoebolavirus sudanense
- espèce Orthoebolavirus taiense
- espèce Orthoebolavirus zairense
- genre Orthomarburgvirus
- genre Striavirus
- espèce Striavirus antennarii
- genre Tapjovirus
- espèce Tapjovirus bothropis
- genre Thamnovirus
- espèce Thamnovirus kanderense
- espèce Thamnovirus percae
- espèce Thamnovirus thamnaconi
Notes et références
Références biologiques
Voir aussi
Wikiwand - on
Seamless Wikipedia browsing. On steroids.




